Chargement de l'article...
Écriture : Romain Tavernier
Relecture scientifique : Anh-Thy Bui
Relecture de forme : Eléonore Pérès et Audrey Denizot
Temps de lecture : environ 11 minutes.
Thématiques : Chimie des matériaux (Chimie)
Publication originale : Martens S., et al., Multifunctional sequence-defined macromolecules for chemical data storage. Nature Communications, 2018. DOI : 10.1038/s41467-018-06926-3
Une équipe de l’université de Gand, en Belgique, a développé une méthode pour encoder un QR code dans des polymères synthétiques. Une simple analyse chimique, dont les résultats sont lus par un programme, permet de restituer l’image du « tag » bien connu. Ces résultats ouvrent la voie vers une utilisation appliquée de ces polymères à très haute définition.
Le stockage d’informations : de l’invention de l’écriture au code-barre moléculaire
Consigner, copier, conserver et transmettre des informations sur un support physique et durable est un processus d’une telle importance pour l’espèce humaine que les historiens ont choisi l’invention de l’écriture pour marquer le début de l’Antiquité. L’invention (ou plutôt le perfectionnement) de l’imprimerie par Gutenberg serait de même corrélée au début de la Renaissance. De nos jours, la révolution numérique est permise par les progrès et la miniaturisation de l’électronique qui permettent de stocker des données dans des espaces toujours plus réduits, et de les transmettre à des vitesses toujours plus élevées.
Pourtant, le stockage d’information sous une forme physique n’est pas une prouesse exclusive à l’espèce humaine. Le monde du vivant repose déjà sur un système très efficace de gestion de l’information. L’ADN, ou acide désoxyribonucléique, est l’une des molécules pilier de l’information génétique. Cette molécule est en réalité assimilable à un système de code, reposant sur quatre bases nucléiques différentes, appelées adénine (A), thymine (T), guanine (G) et cytosine (C) dont l’assemblage dans un ordre précis permet aux systèmes cellulaires de fonctionner correctement, de se multiplier à l’identique, etc. On pourrait imaginer exploiter les possibilités offertes par cet encodage pour des applications à grande échelle, mais la molécule d’ADN est fragile sortie du milieu cellulaire. La chimie de synthèse permet de contourner cette possible limitation en permettant de synthétiser des molécules beaucoup plus stables. De fait, en s’inspirant de l’ADN sur la façon d’intégrer l’information grâce à sa structure moléculaire, on peut élargir l’emploi de molécules contenant des informations. D’autre part, le stockage numérique ne souffre pas d’une telle fragilité de son support. Le disque dur enregistre également ses données sous forme de code, cette fois binaire, en utilisant des 0 et des 1. Cependant, les data centers, qui contiennent les serveurs qui fonctionnent en permanence pour rendre le web disponible à toute heure, consomment beaucoup de place et d’énergie.
Pour imaginer pouvoir s’affranchir des écueils de ces supports d’information tout en conservant leurs avantages, les chimistes ont élaboré de nouvelles méthodes d’encodage. Le stockage de données à l’échelle moléculaire est développé depuis quelques années et les premiers exemples d’applications commencent à fleurir dans la littérature. Ici, l’équipe de Philip Du Prez a élaboré une méthode capable d’encrypter un QR code sur des molécules et de le reconstituer à l’aide de techniques d’analyse chimique très répandues, le tout avec un grand degré d’automatisme.
D’une image en 2D à un assemblage de molécules
Un QR code est cette image en deux dimensions constituée de pixels noirs et blancs, que l’on peut scanner à la manière d’un code barre et qui contient des informations comme du texte ou des liens hypertexte. Alors que les chimistes auraient pu simplement utiliser une série de 0 et de 1 — un langage binaire — pour représenter ces pixels, les auteurs ont fait le choix d’utiliser un langage pentadécimal, c’est-à-dire utilisant quinze caractères différents. En effet, un QR code représente environ 1 089 pixels, soit de façon simplifiée 1 089 caractères binaires (pixels noirs et pixels blancs), alors qu’un langage pentadécimal permet de réduire la même information à environ 410 caractères, ce qui représente un gain de place important [*].
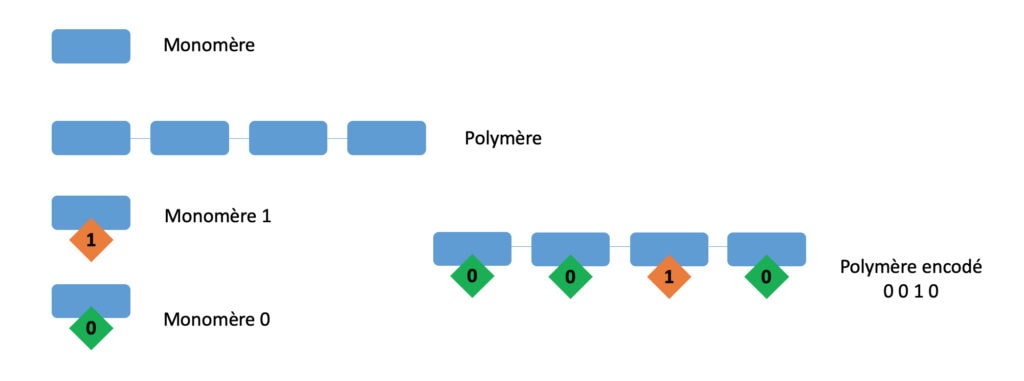
Comment en pratique 410 caractères peuvent-ils être encodés dans des molécules ? S. Martens et ses collaborateurs ont utilisé pour cela des polymères à séquence contrôlée. Un polymère, ou macromolécule, est une grande molécule constituée d’un assemblage d’unités répétées (ou monomères) liées chimiquement. Par exemple, l’amidon, présent dans les céréales, les pommes de terre et les bananes est un polymère naturel composé de l’assemblage de molécules de glucose. Il suffit alors que ces unités moléculaires, les monomères, aient en leur sein un groupement d’atomes qui varie, et l’on pourra assimiler ces variations à des caractères d’encodage différent, comme schématisé dans la Figure 1.
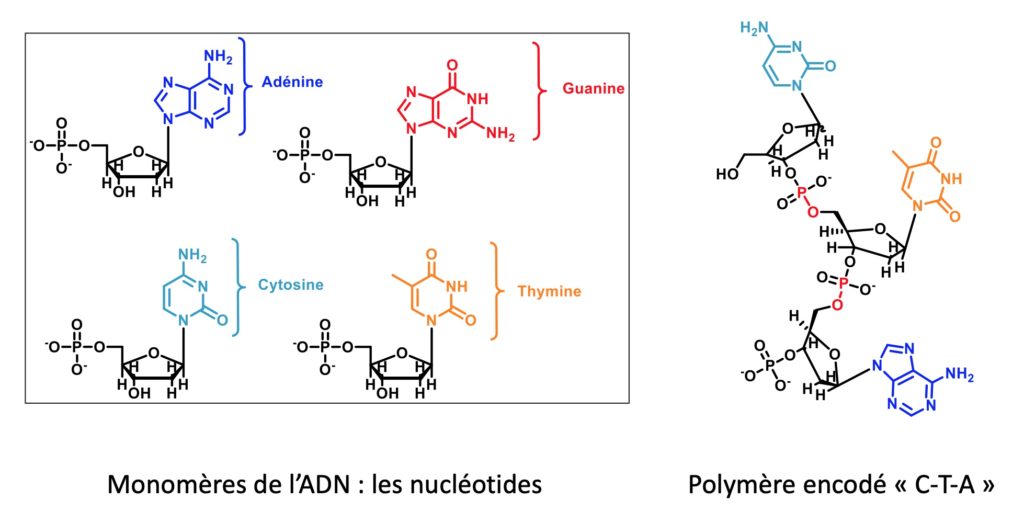
On retrouve ici le même principe que dans l’ADN, qui est en fait un polymère naturel. Les différents monomères de l’ADN sont illustrés dans la Figure 2. Ils ont tous un composant commun qui leur permet de s’assembler (s’attacher ensemble), ainsi que des groupes d’atomes variables : la thymine (T), l’adénine (A), la guanine (G) et la cytosine (C). L’ordre dans lequel les nucléotides de l’ADN s’enchaînent est extrêmement important, définissant par exemple la séquence d’un gène.
Le code pentadécimal utilise 15 unités différentes et représente donc un alphabet de 15 monomères qui, assemblés, forment les polymères encodés. Même avec 410 caractères au lieu de 1 089, on imagine qu’un assemblage de 410 monomères peut être long à définir. Pour optimiser le processus, l’équipe belge a développé un logiciel qui permet, à partir des 1 089 pixels retranscrits sous format numérique en série de 0 et de 1, de définir des assemblages (ou mots) formés à partir des 15 caractères (ou lettres) disponibles. Cela permet l’encodage du QR code, comme illustré schématiquement dans la Figure 3.
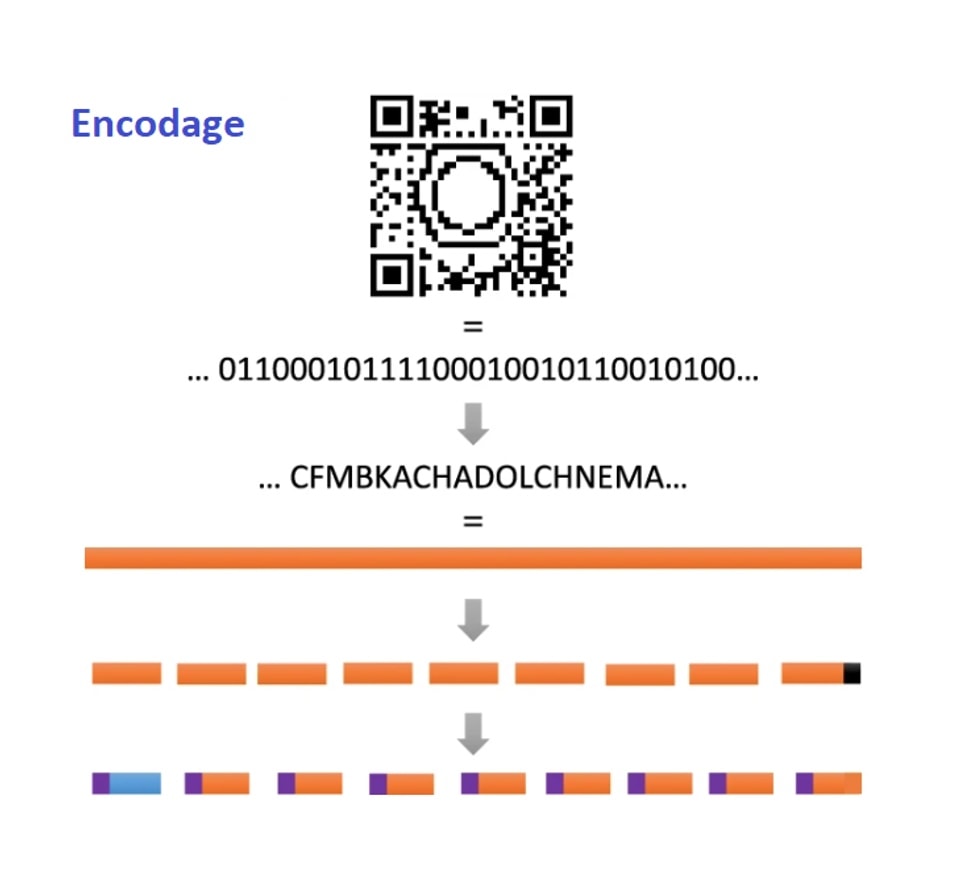
De plus, l’obtention d’un polymère à séquence contrôlée (le polymère encodé) doit se faire en ajoutant chacun des monomères les uns à la suite des autres, ce qui rend le processus de construction en série laborieux. Ainsi, plutôt que de réaliser une seule molécule contenant les 410 caractères du code, il a été divisé en 71 séquences oligomériques, c’est-à-dire 71 assemblages de monomères (les mots) composés de quelques lettres [*]. Cela permet de réaliser les synthèses chimiques de chaque « mot » en parallèle. Chaque séquence contenant également un indicateur numéroté pour indiquer l’ordre des mots dans la phase, il est possible en les décodant de les remettre à leur place, pour former l’image du QR code (Figure 4).
L’automatisme au service de l’encodage et du décodage
Bien que de 1 089 caractères, la tâche des chimistes ait été réduite à 410 monomères assemblés en 71 molécules de quelques unités (les oligomères, petits assemblages de monomères), il n’en reste pas moins que pour synthétiser ces 71 oligomères, il faut soit un peu de temps, soit beaucoup de main d’œuvre. Mais les scientifiques ont pensé à tout, car ils ont spécifiquement choisi des réactions chimiques rapides, efficaces, et surtout automatisables [1]. En effet, l’industrie pharmaceutique a élaboré des synthétiseurs d’ADN permettant de fabriquer ces fameux brins qui contiennent l’information génétique. En utilisant le même type de machine, les chimistes peuvent réaliser la synthèse des 71 oligomères en parallèle, ce qui représente un gain de temps considérable.
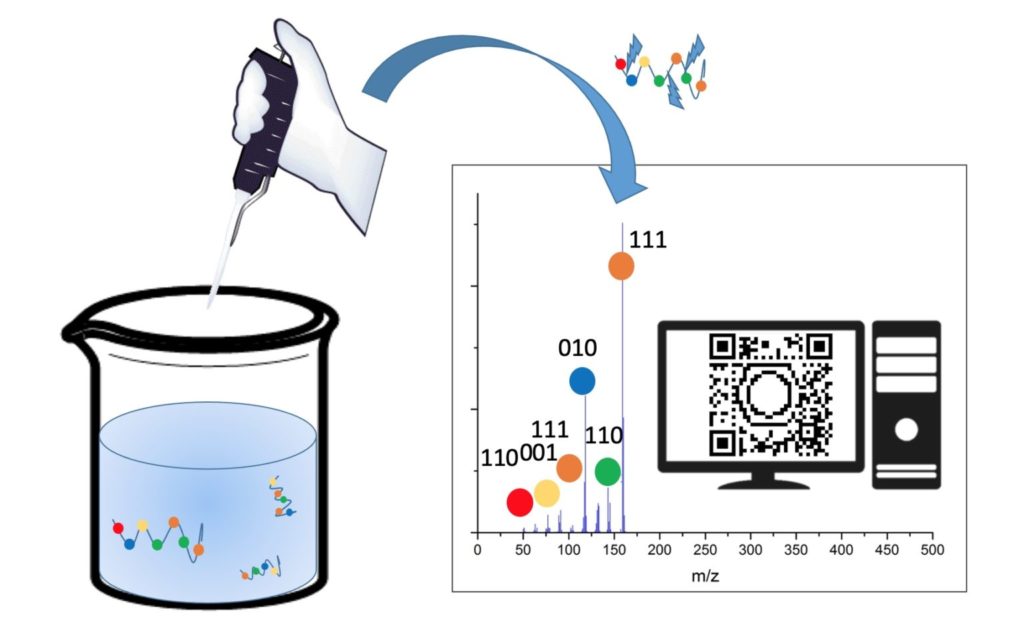
Désormais, il semble très facile de réunir un grand nombre d’informations dans des molécules, et les logiciels nous permettent aisément d’organiser la façon d’encoder l’information dans les polymères. Cependant, qu’en est-il de la lecture de l’information ? Lorsque la synthèse est réussie, comment reconstituer l’information de départ ? S. Martens et ses collègues ont fait le choix d’utiliser une technique qui s’appelle la spectrométrie de masse. Cette méthode possède l’avantage d’être une méthode d’analyse très classique dans les laboratoires de chimie, de biologie ou encore dans la police scientifique. Elle est basée sur la fragmentation des molécules analysées pour en déterminer la masse et permettre d’en déduire sa structure moléculaire. En particulier, comme son nom l’indique, cette technique est basée sur la masse des molécules. Ainsi, pour l’analyse d’un polymère tel qu’élaboré par les scientifiques, l’appareil va simplement découper le polymère en fragmentant un par un les monomères attachés les uns aux autres. La masse moléculaire des fragments et du polymère permettent d’en reconstituer la structure chimique initiale (Figure 4).
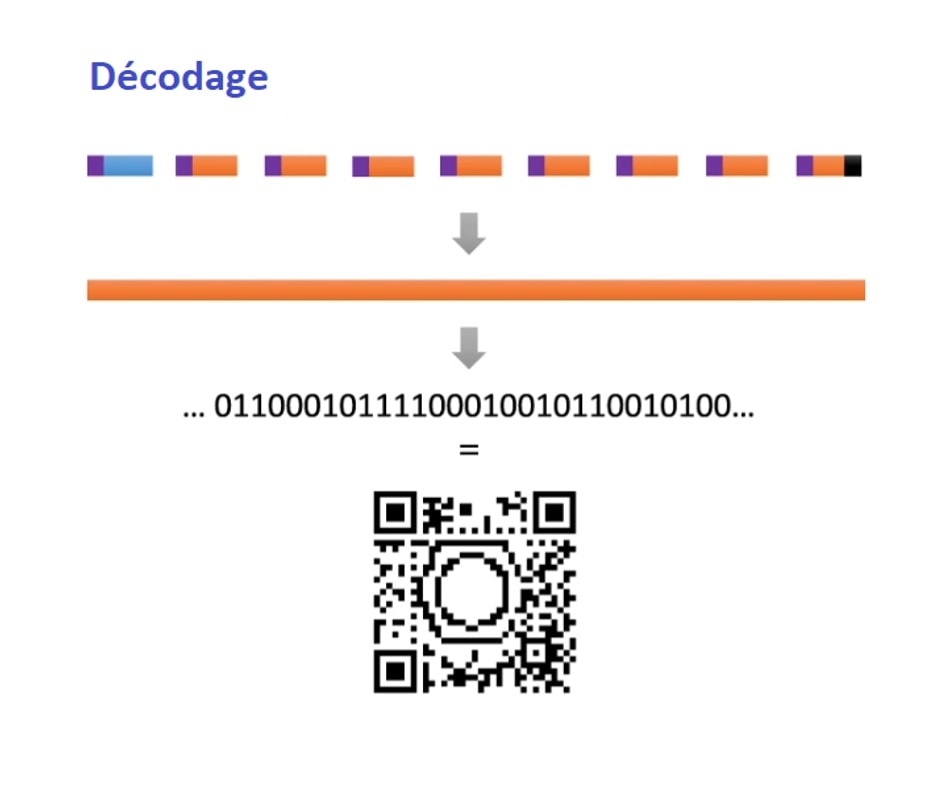
Il est relativement aisé de retrouver la structure du polymère encodé grâce à la spectrométrie de masse. Mais le résultat de l’analyse ne correspond à rien si l’on ne possède pas la clé de lecture. C’est parce que les chimistes ont attribué à ces structures une signification codée que l’on peut en décrypter le message caché, grâce à la grille de lecture. Par exemple, le code morse n’est qu’une succession de points et de traits pour les non initiés. Ainsi, pour le stockage moléculaire, lire un spectre de masse (le résultat de l’analyse) ne permet pas de deviner le code d’un simple coup d’œil. Mais ici encore, l’équipe du professeur Du Prez a pensé à tout. Non seulement l’élaboration du code et la synthèse des molécules codées ont été automatisées, mais c’est également la lecture et la traduction du code qui ont été simplifiées. En effet, un dernier logiciel a été élaboré, grâce auquel on peut également automatiser la traduction du message.
Le décodage du polymère peut donc être réalisé automatiquement. En connaissant la méthode d’encodage, le logiciel peut, à partir de la lecture des analyses des fragments, remonter à l’information encryptée. Le premier logiciel permet de sélectionner la méthode d’encodage adaptée, et le dernier de lire le résultat de spectrométrie de masse, de faire correspondre les résultats aux caractères du code, puis de faire correspondre le code à l’information initiale. Le tout est facilité par le fait que, comme évoqué précédemment, chaque oligomère d’information contient également un indicateur de l’ordre dans lequel reconstituer le code (en violet sur les Figures 3 et 4).
Vers une industrialisation des clés USB moléculaires ?
Les chercheurs ont pu montrer qu’une grande partie des tâches les plus chronophages pour élaborer des polymères codés peut être automatisée, à savoir la définition de la méthode d’encodage et l’attribution des structures de monomères le représentant, la synthèse des polymères effectivement encodés, et la lecture des analyses et leur traduction en information claire. Cependant, une grande partie du travail reste manuelle, comme la synthèse des monomères individuels. Pour l’analyse des oligomères encodés, il faut également les injecter un par un dans la machine. Mais ce travail correspond déjà à une avancée certaine vers des applications concrètes.
Beaucoup de chimistes s’intéressent à ces codes barres moléculaires, et de premiers résultats prometteurs sur des applications de pointe ont déjà été obtenus. En effet, une équipe incluant des chercheurs français a réussi à intégrer, dans un dispositif médical implantable, des polymères contenant une information comme par exemple un numéro de série. Ce dispositif a été implanté avec succès chez des souris, et après quelque temps, explanté pour analyse. Ces polymères n’ont présenté aucune toxicité pour les souris, et surtout, ils ont été décodés avec succès après l’explantation [2]. On peut donc imaginer utiliser un traceur moléculaire pour un dispositif médical implantable, qui serait d’ailleurs difficile à falsifier sans un équipement de laboratoire de chimie.
[*] Il faut noter ici que 1 089 caractères binaires correspondent en réalité à 279 caractères dans un système pentadécimal. Seulement, il est actuellement assez difficile d’obtenir une molécule contenant 279 monomères avec un minimum d’erreurs possibles. Ainsi, les auteurs ont plutôt utilisé plusieurs molécules contenant un petit nombre d’unités pour encoder l’information. Le robot de synthèse automatique ne permettant de réaliser au maximum que 72 synthèses en parallèle, cela a fixé la limite maximale du nombre de séquences autorisées à 71. Les chercheurs ont également dû utiliser des caractères supplémentaires, notamment pour permettre de remettre la séquence dans l’ordre. Cela explique donc l’utilisation de 410 caractères plutôt que 279, pour celles et ceux qui auraient vérifié le calcul.
[1] Smith L. M., Automated Synthesis and Sequence Analysis of Biological Macromolecules. Analytical Chemistry, 1988. DOI : 10.1021/ac00157a001. [Publication scientifique]
[2] Karamessini D., et al., Abiotic Sequence-Coded Oligomers as Efficient In-Vivo Taggants for the Identification of Implanted Materials. Angewandte Chemie International Edition, 2018. DOI : 10.1002/anie.201804895. [Publication scientifique]
Écriture : Romain Tavernier
Relecture scientifique : Anh-Thy Bui
Relecture de forme : Eléonore Pérès et Audrey Denizot
Temps de lecture : environ 14 minutes.
Thématiques : Chimie des matériaux (Chimie)
Publication originale : Martens S., et al., Multifunctional sequence-defined macromolecules for chemical data storage. Nature Communications, 2018. DOI : 10.1038/s41467-018-06926-3

Une équipe de l’université de Gand, en Belgique, a développé une méthode pour encoder un QR code dans des polymères synthétiques. Une simple analyse chimique, dont les résultats sont lus par un programme, permet de restituer l’image du « tag » bien connu. Ces résultats ouvrent la voie vers une utilisation appliquée de ces polymères à très haute définition.
Les polymères à séquence contrôlée : des macromolécules synthétisées avec une précision chirurgicale
Les réactions de polymérisation ont été décrites pour la première fois il y a exactement 100 ans, par Hermann Staudinger, qui recevra pour cela le prix Nobel de chimie en 1953 [*]. Ces réactions conduisent à l’obtention de polymères, ou macromolécules, appelées ainsi car leur taille est bien supérieure aux petites entités moléculaires connues alors. Cela leur confère des propriétés particulières telles que la possibilité de se déformer à chaud ou encore des propriétés d’élasticité inédites dans les matériaux. Depuis ce nouveau paradigme, les applications des polymères ont explosé, et le développement des matières plastiques a connu son essor.
Les réactions de polymérisation ont lieu lorsque des molécules, appelées monomères, s’assemblent les unes avec les autres par une réaction chimique et conduisent à des macromolécules. Par exemple, si la molécule de structure A est capable de subir une polymérisation, on obtiendra des chaînes polymères de structure A-A-A-A-(…). Lorsque plusieurs monomères différents réagissent entre eux, par exemple des molécules de structure A et des molécules de structure B, cette réaction est appelée copolymérisation et donnera par exemple A-B-A-B-A-(…). Les méthodes de polymérisation peuvent conduire à des macromolécules de différentes tailles, et certaines réactions de polymérisation sont anarchiques et incontrôlées. Les polyméristes ont développé de nombreuses méthodes pour contrôler ces réactions, et toutes présentent un niveau de contrôle différent sur la structure des macromolécules. Les polymères à séquence contrôlée résultent des méthodes les plus précises : elles permettent l’insertion d’un monomère à l’endroit voulu dans un enchaînement polymérique.
L’encodage sur polymères : une version synthétique de l’ADN ?
Dans la nature, les polymères de précision peuvent être vecteur d’informations, et ce sans la moindre intervention des talents des polyméristes. Par exemple, l’information véhiculée par l’acide désoxyribonucléique (ADN) repose sur le placement précis de quatre monomères sur une chaîne polymère : les bases de l’ADN. En effet, la majorité des cellules, unités structurelles et fonctionnelles du vivant, contiennent une information génétique stockée dans un espace extrêmement réduit, ce qui signifie qu’un grand nombre d’information est contenu dans un tout petit espace. L’essor du numérique à l’échelle mondiale nous demande de pouvoir stocker toujours plus de données. Aujourd’hui, les disques durs et leur miniaturisation permettent de stocker facilement l’information, de la copier et la transmettre efficacement. Mais les data centers, qui contiennent les serveurs nécessaires pour faire fonctionner notre environnement numérique, sont très énergivores. Malgré ces avancées en termes de miniaturisation, un stockage à l’échelle moléculaire comme celui de l’ADN présente des avantages difficilement atteignables par la technologie utilisant le principe du disque dur. En effet, un tel stockage moléculaire permet d’utiliser des éléments chimiques abondants (le carbone, l’oxygène, l’hydrogène et l’azote, qui forment ensemble des composés organiques). De plus, cela permet un stockage compact de l’information. Le numérique utilisant une base 2 (0 ou 1) et l’ADN une base 4 (les quatre bases de l’ADN), les informations sont encodées avec une combinaison de deux ou quatre caractères, respectivement. Pourtant, si l’ADN est un support performant pour véhiculer l’information biologique, il l’est moins sorti du milieu physiologique. En effet, la molécule d’ADN est fragile et résiste peu aux agressions chimiques et à la température. Ces inconvénients limiteraient donc son utilisation pour stocker des données à notre échelle. Dans le cas des polymères, on peut utiliser n’importe quelle base qui sera adaptée à l’information devant être stockée. En choisissant bien les polymères synthétiques que l’on utilisera, on peut donc adapter ce support d’informations à des applications variées, par exemple des polymères biocompatibles pour applications biomédicales.
En pratique, comment les chimistes pourraient-ils combiner les avantages des polymères de précision comme l’ADN, tout en évitant les inconvénients de ce dernier ? Les polymères synthétiques peuvent avantageusement être conçus pour résister à des conditions de stockage données. Cependant, il ne suffit pas de prendre n’importe quel polymère pour le transformer en disque dur miniature. En effet, afin de pouvoir utiliser un polymère pour stocker de l’information, encore faut-il pouvoir le décoder. Les biologistes ont par exemple mis au point des méthodes de séquençage permettant de « décoder » l’ADN. Les polymères synthétiques présentent un avantage : on peut décider en amont de la méthode d’encodage de l’information, de la méthode de séquençage de l’information et des structures chimiques qui conviendront à tout cela à la fois.
Pour pouvoir « décoder » les polymères à séquence contrôlée synthétiques, une méthode d’analyse en particulier est extrêmement adaptée : la spectrométrie de masse. Elle permet l’identification précise des structures moléculaires grâce à leur masse moléculaire. Cette technique est très répandue dans les laboratoires d’analyses chimiques car rapide et simple à mettre en place. Deux actions permettent l’élucidation de la structure moléculaire. Tout d’abord, l’instrument fragmente les molécules analysées. Un fragment est alors un « morceau » de molécule, une molécule plus petite issue de la première entité, dans laquelle quelques liaisons chimiques se sont rompues. La séquence préalablement codée par les chercheurs est construite de sorte que les fragments correspondent à une unité codante, ou à un ensemble d’unités codantes. Ces fragments sont ensuite « pesés » pour déterminer leur masse avec une précision à l’atome près, permettant de retrouver la structure moléculaire des fragments. À la fin, le réassemblage (mathématique) des masses des fragments permet de remonter à la séquence initiale, et donc à l’information inscrite [**].
Écrire sur des polymères : la méthode
En pratique, il faut de nombreux outils pour construire une séquence polymère encodée. Il faut d’abord choisir la méthode de synthèse du polymère pour garder un contrôle parfait du placement des unités. Pour cela, S. Martens et ses collègues, dont la publication nous intéresse ici, ont utilisé la synthèse en phase solide, qui permet de faire croître des brins de polymères, unité par unité, à partir de billes de polystyrène [***]. Cette méthode a fait ses preuves pour la synthèse de peptides, ces polymères d’acides aminés qui ont de nombreuses fonctions dans le vivant.
Afin de pouvoir distinguer deux unités codantes différentes, il faut que ces unités aient un signal différent lorsqu’elles sont analysées en spectrométrie de masse. Les chercheurs analysent pour cela la masse moléculaire, qui permet de distinguer chaque unité. Une masse moléculaire donnée représente alors une unité codante particulière. Pour mettre ce principe en pratique, il suffit de faire varier des groupements chimiques, qui seront la partie « codante » du monomère. Chaque monomère est ainsi construit sur la même base : un squelette qui présente la réactivité chimique permettant la bonne construction de la séquence, et la partie variable codante.
Cependant, un polymère ne peut posséder un nombre d’unités infini. De plus, plus la chaîne polymère contient un grand nombre d’unités, plus elle est susceptible de contenir des erreurs. Dans notre cas, le risque serait alors de mal décrypter le message. Il est donc préférable de découper le code complet en petites séquences. Une phrase sera par exemple encodée en utilisant un polymère par mot. Ces petites séquences polymères comprenant un nombre d’unités relativement faible, sont appelées oligomères.
Enfin, pour décoder une phrase, encore faut-il que les mots soient dans le bon ordre. Pour cela, rien de plus simple, il suffit d’ajouter à la séquence une unité qui permet de restituer l’ordre des oligomères dans la séquence complète.
Pour vérifier la robustesse de la méthode de codage et de décodage, les chimistes se sont attelés à encoder « To write or not to write on oligos? » (« Écrire ou ne pas écrire sur des oligos ? »). Ils ont d’abord attribué un numéro à chacun des huit mots constituant la séquence, pour ordonner les séquences. Ils ont synthétisé au total 19 monomères de masses différentes permettant l’encodage des lettres, des 8 indicateurs de position ainsi que le point d’interrogation. Les 8 séquences ont été synthétisées à la main, étape par étape, donc monomère par monomère. Les oligomères codés, dont deux exemples sont illustrés Figure 1, ont ensuite été analysés par spectrométrie de masse, séparément, et leurs spectres ont permis de reconstituer la phrase de départ.
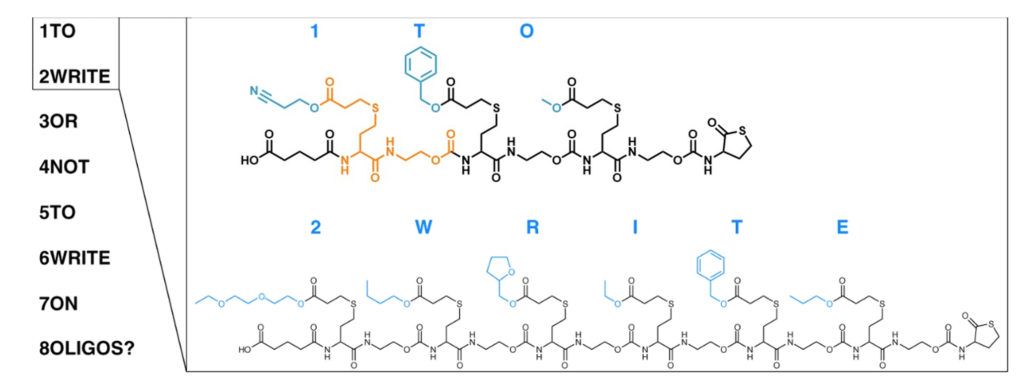
Pour comprendre la phrase, encore faut-il connaître la façon d’encoder les lettres (la grille de traduction) et la taille de chacune des séquences. Plus précisément, le spectre de masse permet d’abord de retrouver la masse d’un oligomère, puis la fragmentation sépare l’oligomère en fragments correspondant à un ou plusieurs monomères. La combinaison des différents fragments permet ensuite de reconstituer la séquence de monomères. Ainsi, dans l’exemple présenté dans la Figure 1, le spectre de masse peut donner la masse correspondant à la séquence 2WRITE, mais aussi les fragments 2WRIT*, 2WRI*, 2WR* et 2*. Le premier chiffre permet d’indiquer la place de l’oligomère dans le code complet. Ainsi, on obtient un ordre dans lequel placer le code contenu dans chacun des oligomères analysés.
Les robots et les algorithmes au service de l’encodage chimique
En 2012, un article dans la revue Science décrivait l’utilisation de l’ADN comme support d’écriture pour y inscrire un texte de plus de 50 000 mots et 11 images grâce à plus de 50 000 oligonucléotides [1]. Pour synthétiser ces 50 000 oligonucléotides, on pourrait penser que cette méthode impliquerait le recrutement d’une équipe de chimistes travaillant 24h/24.
Qu’en serait-il pour encoder un QR code ? Précisons que le QR code est un arrangement en 2D de 1 089 pixels blancs et noirs, chaque pixel étant en fait un bit d’information 0 ou 1. Cette méthode d’encodage peut être très performante. En effet, les réactions chimiques entre les différents monomères ainsi que l’analyse par spectrométrie de masse se produisent en quelques minutes (utilisation de larges excès de molécules très réactives). Les étapes les plus chronophages du processus complet sont l’élaboration de la méthode d’encodage, la traduction de l’information en code, la synthèse manuelle des séquences codantes et la lecture des analyses permettant de remonter à l’information.
Encore une fois, Steven Martens et ses collègues ont élaboré une méthode pour optimiser toutes ces étapes. Pour le choix de l’encodage, l’équipe de chimistes a développé un algorithme qui transforme un code (binaire par exemple) en séquences de monomères différents et propose les structures chimiques à synthétiser. La synthèse manuelle a été remplacée par un robot. En effet, la synthèse en phase solide a largement démontré sa capacité à être automatisée [2]. Chaque ajout d’une unité codante est ainsi réalisée dans des petits réacteurs par des bras robotisés, permettant de nombreuses synthèses parallèles (comme cela existe d’ailleurs pour la synthèse d’ADN [3]). Enfin, les données de sortie de l’analyse par spectrométrie de masse sont les masses des fragments des oligomères. Ces données numériques ont également pu être analysées par un algorithme développé pour l’occasion. En ayant pour paramètres d’entrée les différentes structures utilisées pour encoder (leur masse) ainsi que les caractères correspondants, il devient alors très simple de déchiffrer l’information analysée.
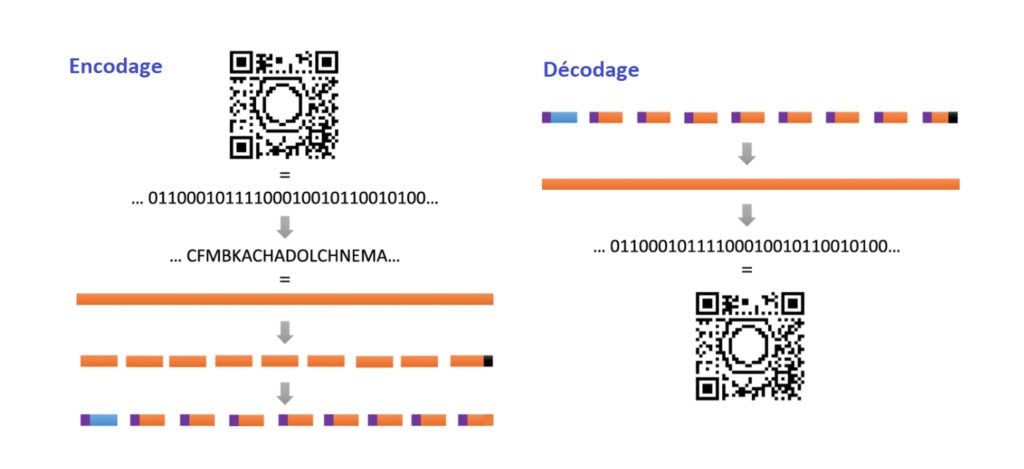
Pour l’occasion, l’équipe a démontré qu’il était possible d’encoder un QR code, en utilisant, plutôt qu’un langage binaire, un langage pentadécimal utilisant les 15 premières lettres de l’alphabet (Figure 2). Les 1 089 bits d’informations ont été traduits grâce à l’algorithme d’encodage en 71 séquences, toutes de tailles égales, dont le nombre total est ajustable. L’utilisation de séquences de nombre d’unités égal permet de faciliter la lecture finale car c’est un paramètre que le logiciel de décryptage doit prendre en compte. S’il y en a un de moins, l’algorithme est plus rapide.
Un robot a pu réaliser la synthèse des 71 séquences en parallèle [****]. Les 71 résultats d’analyse par spectrométrie de masse ont ensuite été déchiffrés par le second algorithme, remontant in fine au QR code sous forme de bits d’information.
L’équipe de l’université de Gand a ainsi choisi d’encoder le QR code représentant un lien vers la page Wikipédia d’Auguste Kékulé, le chimiste ayant découvert la structure chimique du benzène lors de son passage dans cette même université, en 1865. D’ailleurs, lors de l’élaboration du QR code, les chimistes y ont inclus au centre la représentation schématique du benzène : un hexagone, représentant un cycle comprenant six atomes de carbone, contenant en son centre un cercle, représentant les doubles liaisons entre les atomes.
Les travaux présentés dans cet article montrent que, malgré la complexité apparente de coder des informations sur des molécules, les outils de synthèse et d’analyse existant aujourd’hui permettent de rendre le concept quasiment applicable à une grande échelle. De plus, le concept présenté dans la publication permet d’illustrer la possibilité d’insérer des informations complexes, comme ici un QR code. Les travaux d’encodage sur des polymères à séquence contrôlée illustrent également les avancées de ce domaine, qui consistent à contrôler parfaitement la structure chimique des macromolécules, pour des applications de pointe, notamment dans le domaine biomédical avec le traçage de dispositifs implantables. En effet, il a été montré qu’il était possible de disperser des polymères synthétiques encodés dans un dispositif biocompatible implanté dans des souris. Après explantation, il a été possible d’identifier le code dispersé dans le dispositif. Ce traçage est invisible et peut éviter la contrefaçon, ou l’effacement d’un marquage physique par usure [4]. Néanmoins, il faudra probablement encore adapter les outils utilisés à ceux qui existent déjà dans l’industrie, de nombreuses opérations étant encore réalisées à la main et à petite échelle.
[*] Le prix Nobel de Chimie 1953 a été attribué à Hermann Staudinger « pour ses découvertes dans le domaine de la chimie macromoléculaire. »
[**] Pour être exact, l’équipe a utilisé la spectrométrie de masse tandem. Les analytes sont fragmentés une première fois, puis chaque fragment est de nouveau fragmenté, avec des conditions de fragmentation différentes. Cela permet d’obtenir beaucoup plus d’information à partir d’une même analyse.
[***] Robert Bruce Merrifield reçoit en 1984 le prix Nobel de chimie pour le développement de la synthèse en phase solide. Les billes de polystyrène utilisées pour la synthèse sont également appelées « résines de Merrifield ».
[****] Il faut noter ici que 1 089 caractères binaires correspondent en réalité à 279 caractères dans un système pentadécimal. Seulement, il est actuellement assez difficile d’obtenir une molécule contenant 279 monomères avec un minimum d’erreurs possibles. Ainsi, les auteurs ont plutôt utilisé plusieurs molécules contenant un petit nombre d’unités pour encoder l’information. Le robot de synthèse automatique ne permettant de réaliser au maximum que 72 synthèses en parallèle, cela a fixé la limite maximale du nombre de séquences autorisées à 71. Les chercheurs ont également dû utiliser des caractères supplémentaires, notamment pour permettre de remettre la séquence dans l’ordre. Cela explique donc l’utilisation de 410 caractères plutôt que 279, pour celles et ceux qui auraient vérifié le calcul.
[1] Church G. M., et al., Next-Generation Digital Information Storage in DNA. Science, 2012. DOI : 10.1126/science.1226355. [Publication scientifique]
[2] Chan W. C. & White P. D., « Fmoc Solid Phase Peptide Synthesis, A Practical Approach » (Chapitre 13). Oxford University Press, 2000. [Livre de science]
[3] Smith L. M., Automated Synthesis and Sequence Analysis of Biological Macromolecules. Analytical Chemistry, 1988. DOI : 10.1021/ac00157a001. [Publication scientifique]
[4] Karamessini, D., et al., Abiotic Sequence-Coded Oligomers as Efficient In-Vivo Taggants for the Identification of Implanted Materials. Angewandte Chemie International Edition, 2018. DOI : 10.1002/anie.201804895. [Publication scientifique]