Chargement de l'article...
Écriture : Margaux Demuysere
Relecture scientifique : Sylvie Manuse et Anaïs Biclot
Relecture de forme : Fanny Grisetto et Pierre Marrec
Temps de lecture : environ 11 minutes.
Thématiques : Microbiologie (Biologie)
Publication originale : Hill P., et al., The vulnerable versatility of Salmonella antibiotic persisters during infection, Cell Host & Microbe, 2021. DOI : 10.1016/j.chom.2021.10.002

Malgré des antibiotiques efficaces, il arrive que certaines infections bactériennes rechutent. Un casse-tête médical sur lequel planchent les scientifiques depuis plusieurs années déjà. Dans cette publication, Peter Hill et ses collègues étudient la population d’irréductibles bactéries dont les patients atteints de salmonellose invasive récurrente n’arrivent pas à se débarrasser. L’équipe met également en lumière un ingrédient de la potion magique nécessaire à ce tour de force bactérien.
Résister, survivre ou périr
Nous sommes en l’an 95 après la découverte de la pénicilline, l’un des premiers antibiotiques utilisés en clinique, par Alexander Fleming [*]. Les épidémies de peste et de choléra ne sont plus qu’un effrayant souvenir, toutes les bactéries ont rendu les armes. Toutes ? Non ! Certaines bactéries résistent et persistent encore et toujours malgré les antibiotiques. Et la vie n’est pas facile pour les patients en échec thérapeutique.
Avant de scruter ces bactéries récalcitrantes au microscope, revenons un peu sur l’ennemi mortel de ces microbes : les antibiotiques. Les antibiotiques sont des molécules capables de tuer les bactéries ou de les empêcher de se multiplier.
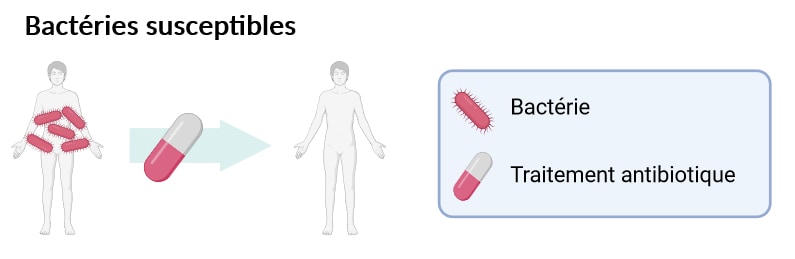
C’est seulement au début du 20ème siècle que les scientifiques découvrent l’existence d’antibiotiques dans la nature et commencent à les exploiter afin de traiter les infections bactériennes [1]. C’est une véritable révolution médicale ! Mais tandis que les bactéries dites susceptibles meurent sous l’action des antibiotiques (Figure 1), l’apparition de bactéries dites résistantes est rapidement signalée [2].
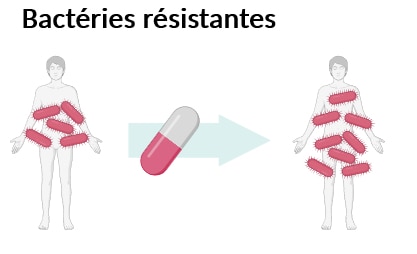
Une bactérie est qualifiée de résistante lorsqu’elle parvient à proliférer, c’est-à-dire à se multiplier, malgré la présence d’un ou plusieurs antibiotiques (Figure 2). Différentes stratégies leur permettent en effet de se soustraire à l’action d’un antibiotique. Elles peuvent par exemple le détruire, l’expulser hors de leur système avec un système de pompes, ou encore modifier la cible de l’antibiotique qui ne peut alors plus la reconnaître. Le traitement administré est donc conditionné par l’arsenal des mécanismes de défense dont dispose une bactérie donnée. L’antibiorésistance peut rendre difficile voire impossible le traitement de certaines infections multi-résistantes, et a entraîné la mort de plus d’un million de personnes dans le monde rien qu’en 2019, selon de récentes estimations [3].
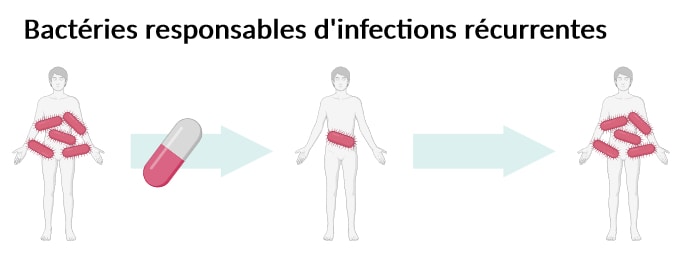
Si l’antibiorésistance est un énorme problème de santé publique, elle n’est pas toujours à l’origine de l’échec d’un traitement. En effet, certaines infections ont tendance à rechuter après un traitement antibiotique pourtant jugé efficace. C’est-à-dire que les bactéries responsables de l’infection, pourtant susceptibles à l’antibiotique administré et donc censées être tuées par celui-ci, sont capables de survivre et de recommencer à proliférer après l’arrêt du traitement. (Figure 3). Le patient alterne alors entre périodes de rémission et de rechute, entrecoupées de prises d’antibiotiques. Ce phénomène de récurrence concerne notamment certaines infections urinaires, ostéo-articulaires, ou encore la tuberculose [4].
Deux raisons peuvent expliquer l’échec d’un antibiotique à éradiquer complètement les bactéries responsables d’une infection. Premièrement, si la dose d’antibiotique administrée est insuffisante. Cela peut par exemple se produire si le patient arrête de prendre ses médicaments avant la fin du traitement. Deuxièmement, si les bactéries mettent en place des stratégies de survie durant la durée du traitement. Cette situation se distingue de la résistance car ici les bactéries ne sont pas capables de proliférer en présence de l’antibiotique. En revanche, elles parviennent à se maintenir en vie durant la durée du traitement via des mécanismes encore mal compris.
Par Toutatis, qui sont ces bactéries qui trompent la mort ? C’est ce que l’équipe de la docteure Sophie Helaine s’attelle à étudier.
D’irréductibles salmonelles
Dans cette publication, l’équipe de recherche s’intéresse à la bactérie Salmonella enterica, principale responsable de la salmonellose. La salmonellose est une infection du tube digestif qui entraîne le plus souvent une gastro-entérite, mais qui peut également devenir invasive en s’étendant à d’autres organes. Chez les patients immunodéprimés [**], les salmonelloses invasives ont tendance à récidiver après traitement [5, 6]. C’est ce modèle qui est choisi afin d’étudier les bactéries responsables d’infections récurrentes.
Pour comprendre comment les salmonelles échappent à l’extermination totale durant un traitement antibiotique, l’équipe entreprend de récapituler en laboratoire ce qui se joue à l’intérieur des patients. Pour ce faire, Peter Hill et ses collègues utilisent des bactéries directement collectées chez des patients souffrant de salmonellose invasive récurrente [***] pour infecter des cellules immunitaires de souris, appelées macrophages. En effet, les macrophages sont des cellules immunitaires présentes dans tous les organes et notamment au niveau des intestins qui sont une cible privilégiée des salmonelles.
En temps normal, les macrophages assurent la défense de la muqueuse en phagocytant (= en avalant) les bactéries qui se trouvent dans leur périmètre de surveillance. Les bactéries phagocytées sont alors détruites, et les macrophages alertent le reste du système immunitaire de la détection d’un intrus. Lors de certaines infections, les macrophages échouent dans leur rôle de défense et deviennent au contraire des réservoirs à bactéries. En effet, les bactéries phagocytées échappent à la digestion par les macrophages, et les utilisent alors comme abri pour proliférer tout en se cachant de la réponse immunitaire. Une fois cette dernière calmée, elles sont alors capables de ré-initier une infection [7].
Dans le cadre d’infections à salmonelles invasives récurrentes, les macrophages sont justement soupçonnés d’abriter les bactéries dissidentes responsables des rechutes [8]. C’est pourquoi l’équipe veut évaluer l’efficacité des antibiotiques contre ces bactéries dissimulées dans les macrophages.
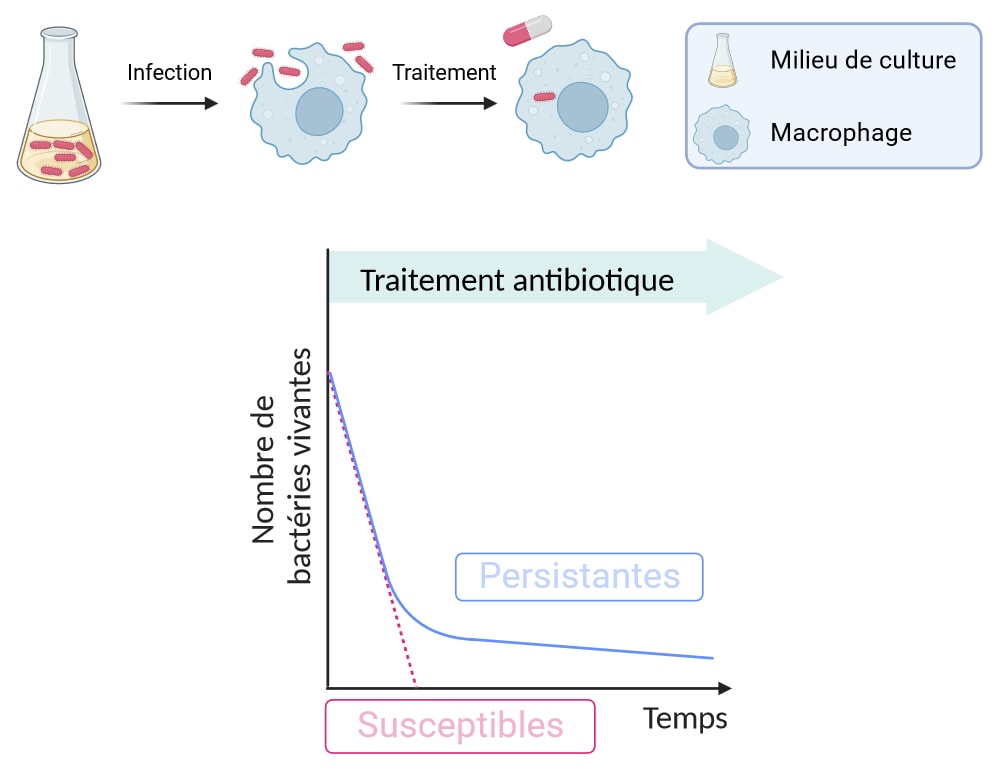
Les chercheurs infectent donc des macrophages de souris en les mettant en contact avec des salmonelles de patients qu’ils vont phagocyter. Ils ajoutent ensuite un antibiotique et suivent la survie des bactéries au cours du temps (Figure 4). Les courbes de survie obtenues pour les échantillons testés (Figure 4, courbe bleue) sont comparées à la courbe théoriquement attendue pour des bactéries susceptibles à l’antibiotique (Figure 4, courbe rose pointillée). Contrairement à cette dernière, les courbes de survie des salmonelles isolées chez les quatre patients testés présentent deux phases : une première phase de mort rapide, semblable au comportement de bactéries susceptibles, et une deuxième phase de plateau, indiquant la survie d’une minorité de la population.
Ces courbes de survie biphasiques sont caractéristiques de la présence de bactéries qualifiées de persistantes. Les bactéries persistantes sont capables de survivre de façon prolongée à un stress antibiotique, tandis que le reste de la population bactérienne, pourtant génétiquement identique et à laquelle elles appartiennent, meurt. Un peu comme un certain village d’irréductibles Gaulois qui s’oppose à Jules César tandis que le reste de la Gaule tombe sous sa coupe.
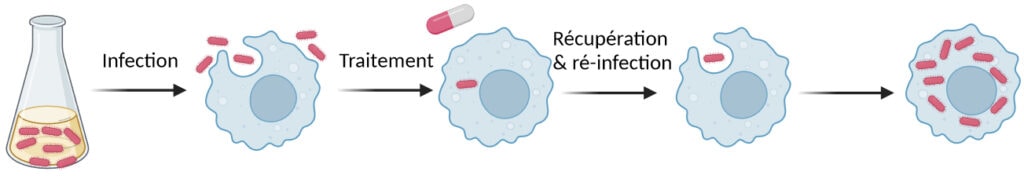
Ces irréductibles salmonelles sont-elles capables de faire rechuter une infection après l’arrêt du traitement antibiotique ? Au laboratoire, la réponse est oui ! Les bactéries persistantes ayant survécu au traitement antibiotique sont capables d’infecter de nouveaux macrophages de souris après avoir été phagocytées (Figure 5). Cela en fait des coupables idéales pour la recrudescence des salmonelloses invasives.
Un ingrédient de la potion magique
Reste à comprendre le secret de la survie de ces bactéries persistantes malgré la présence d’antibiotiques… Pour cela, pas besoin de torturer Panoramix ! Les chercheurs utilisent une technique de séquençage d’ARN messagers (ARNm) pour espionner les bactéries persistantes.
Les ARNm sont des molécules contenant des instructions pour la fabrication de protéines. Séquencer les ARNm permet de savoir quelles protéines sont fabriquées par la bactérie, et renseigne indirectement sur son environnement. Par exemple, si les bactéries produisent des protéines permettant de digérer le lactose, cela signifie probablement qu’elles se trouvent dans un milieu riche en lactose.
Les bactéries persistantes, toujours en vie au sein des macrophages après le traitement antibiotique, sont isolées et leur ARNm est séquencé. Cette analyse montre que les bactéries persistantes produisent des ARNm instruisant la fabrication de protéines participant à la réponse SOS. Cette réponse, qui porte bien son nom, tire la sonnette d’alarme chez les bactéries lorsque l’ADN est endommagé et présente des cassures. Elle entraîne alors la production de plusieurs protéines permettant de réparer les lésions de l’ADN.
La réponse SOS étant uniquement active lorsque les bactéries présentent des lésions de l’ADN, l’équipe investigue l’état de l’ADN des bactéries persistantes. Grâce à une technique qui rend les extrémités cassées de l’ADN fluorescentes, les chercheurs montrent que les bactéries persistantes présentent effectivement des cassures de l’ADN lorsqu’elles sont dans les macrophages traités aux antibiotiques. En creusant encore, l’équipe de recherche montre que les cassures sont causées par les macrophages, de par l’activité de leurs défenses immunitaires. L’induction de la réponse SOS par les bactéries persistantes leur permet donc de réparer leur ADN, attaqué par les macrophages au sein desquels elles se multiplient (Figure 6, panel de gauche).
Arrêtez ce Druide, par Neptune !
La découverte de l’importance du système SOS pour la survie des bactéries persistantes représente-t-elle un potentiel talon d’Achille pour écraser une fois pour toutes ces irréductibles salmonelles ?
La réponse SOS induit notamment la production d’une protéine, appelée RecA, importante pour réparer les lésions de l’ADN. L’équipe de recherche montre qu’empêcher la production de RecA impacte négativement le taux de survie et la capacité des bactéries survivantes à recommencer leur prolifération après l’arrêt du traitement (Figure 6, panel de droite).
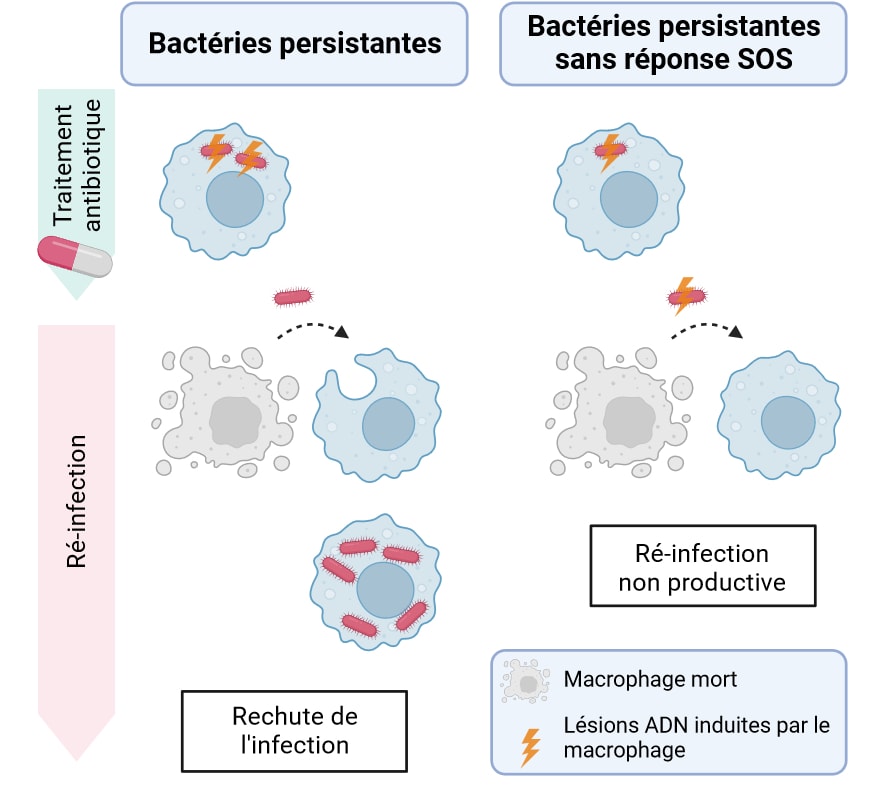
Les chercheurs ont également testé l’importance de leur découverte dans un modèle animal de salmonellose invasive récurrente. Des souris sont infectées par des salmonelles dépourvues de RecA. À la fin du traitement antibiotique, il reste beaucoup moins de bactéries vivantes dans les organes que lorsqu’on infecte les souris avec des bactéries non modifiées. Plus prometteur encore, il n’y a eu aucune rechute de l’infection chez les souris infectées avec les bactéries sans système de réparation, au contraire des souris infectées par les bactéries non modifiées. Le blocage de la réponse SOS serait-il la clé pour permettre aux antibiotiques de venir à bout des salmonelloses qui rechutent ? Espérons une réponse dans les prochains tomes.
[*] Alexander Fleming a découvert la pénicilline en 1928 et publié ses observations en 1929 [9]. Pour en savoir plus sur l’histoire des antibiotiques, vous pouvez également consulter cet article [1], ou écouter l’épisode 50 du podcast “This Podcast Will Kill You” (en anglais).
[**] On qualifie un patient d’immunodéprimé lorsque son système immunitaire est affaibli, ou moins efficace que celui de la population générale. Cela peut être dû entre autres à une anomalie génétique, à un cancer ou à une infection, notamment par le VIH (virus d’immunodéficience humaine). Les patients immunodéprimés sont plus susceptibles aux infections et au développement de cancers.
[***] Ces échantillons ont été collectés lors d’une étude clinique précédente, réalisée par une équipe indépendante [5].
[1] Gould K., Antibiotics: from prehistory to the present day. Journal of Antimicrobial Chemotherapy, 2016. DOI : 10.1093/jac/dkv484. [Review]
[2] Fisher R.A., et al., Persistent bacterial infections and persister cells. Nature Reviews Microbiology, 2017. DOI : 10.1038/nrmicro.2017.42. [Review]
[3] Murray C.J.L., et al., Global burden of bacterial antimicrobial resistance in 2019: a systematic analysis. The Lancet, 2022. DOI : 10.1016/S0140-6736(21)02724-0. [Publication scientifique]
[4] Abraham E.P. & Chain E., An Enzyme from Bacteria able to Destroy Penicillin. Nature, 1940. DOI : 10.1038/146837a0. [Publication scientifique]
[5] Okoro C.K., et al., High-resolution single nucleotide polymorphism analysis distinguishes recrudescence and reinfection in recurrent invasive nontyphoidal Salmonella typhimurium disease. Clinical Infectious Diseases: An Official Publication of the Infectious Diseases Society of America, 2012. DOI : 10.1093/cid/cir1032. [Publication scientifique]
[6] Organisation Mondiale de la Santé, Salmonella (infections à, non typhiques). OMS, 2018. [Site internet, consulté le 13/05/2023]
[7] Price J.V. & Vance R.E., The macrophage paradox. Immunity, 2014. DOI : 10.1016/j.immuni.2014.10.015. [Review]
[8] Gordon M.A., et al., Invasive Non-typhoid Salmonellae Establish Systemic Intracellular Infection in HIV-Infected Adults: An Emerging Disease Pathogenesis. Clinical Infectious Diseases, 2010. DOI : 10.1086/651080. [Publication scientifique]
[9] Fleming A., On the Antibacterial Action of Cultures of a Penicillium, with Special Reference to Their Use in the Isolation of B. Influenzæ. British Journal of Experimental Pathology, 1929. PMCID : PMC2048009. [Publication scientifique]
Écriture : Margaux Demuysere
Relecture scientifique : Sylvie Manuse et Anaïs Biclot
Relecture de forme : Fanny Grisetto et Pierre Marrec
Temps de lecture : environ 17 minutes.
Thématiques : Microbiologie (Biologie)
Publication originale : Hill P., et al., The vulnerable versatility of Salmonella antibiotic persisters during infection, Cell Host & Microbe, 2021. DOI : 10.1016/j.chom.2021.10.002

Malgré des antibiotiques efficaces, il arrive que certaines infections bactériennes rechutent. Un casse-tête médical sur lequel planchent les scientifiques depuis plusieurs années déjà. Dans cette publication, Peter Hill et ses collègues étudient la population d’irréductibles bactéries dont les patients atteints de salmonellose invasive récurrente n’arrivent pas à se débarrasser. L’équipe met également en lumière un ingrédient de la potion magique nécessaire à ce tour de force bac
Les adversaires de César
Nous sommes en l’an 95 après la découverte de la pénicilline par Alexander Fleming [*]. Les épidémies de peste et de choléra ne sont plus qu’un effrayant souvenir, toutes les bactéries ont rendu les armes. Toutes ? Non ! Depuis longtemps déjà, l’existence de poches de résistance est signalée, notamment avec l’observation de bactéries capables de dégrader la pénicilline [1].
Si les enjeux vitaux de l’antibiorésistance font régulièrement leur apparition dans les médias, d’autres bactéries récalcitrantes aux antibiotiques préoccupent également le personnel soignant. Ces bactéries, bien que susceptibles à l’antibiotique administré et donc censées être tuées par celui-ci, sont capables de ré-initier une infection à la suite de l’arrêt du traitement. Un cycle infernal de traitement-rechute commence alors, un véritable cauchemar pour les patients. Ces infections récurrentes concernent entre autres certaines infections urinaires, ostéo-articulaires, ou encore la tuberculose [2].
Par Toutatis, qui sont ces bactéries qui trompent la mort ?! Deux phénotypes étudiés au laboratoire par les microbiologistes pourraient expliquer cette rechute en dépit d’un traitement apparemment efficace : la tolérance et la persistance.
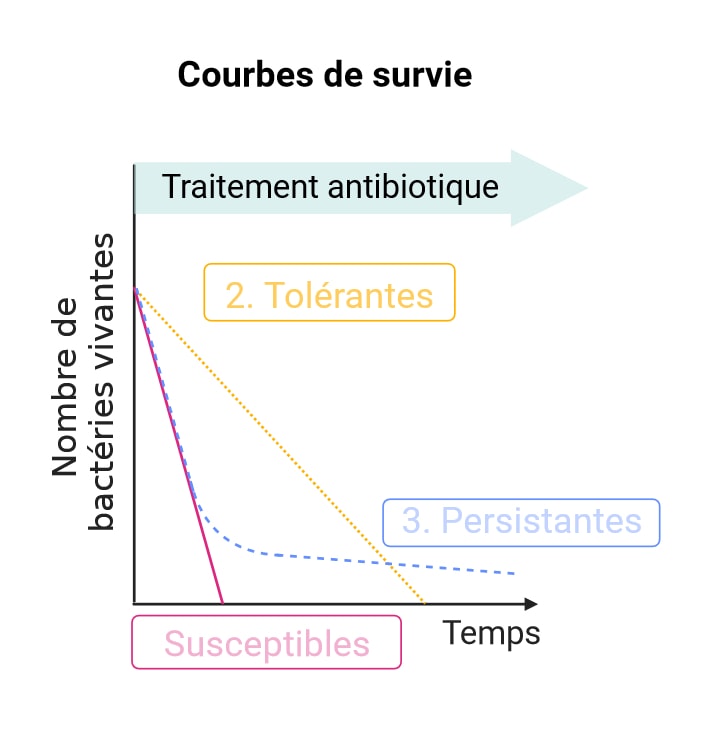
Les bactéries tolérantes sont semblables à des ours en hibernation. Comme eux, elles fonctionnent à métabolisme ralenti. Cela les rend moins susceptibles aux antibiotiques, davantage actifs contre les bactéries en croissance. Cette efficacité amoindrie se traduit par une mort plus lente des bactéries tolérantes par rapport aux bactéries susceptibles. Une mort lente pour une durée de traitement antibiotique standard… Voilà qui pourrait permettre à une partie des bactéries tolérantes encore en vie à la fin du traitement de connaître un nouveau printemps !
Quant aux bactéries persistantes, elles s’apparentent davantage à un village d’irréductibles Gaulois qui lutte encore et toujours contre l’envahisseur. En effet, l’antibiotique est capable de tuer l’entièreté de la population, sauf une poignée de bactéries, qualifiées de persistantes. Ces dernières sont capables de survivre sans se diviser durant la durée du traitement et de recommencer à proliférer une fois l’antibiotique arrêté. Les mécanismes responsables de la formation de bactéries persistantes, ainsi que de leur survie, sont encore élusifs et probablement multiples. En effet, certaines bactéries persistantes semblent dormantes (de façon similaire aux bactéries tolérantes), tandis que d’autres maintiennent leur activité métabolique et peuvent même influencer la réponse immunitaire.
Contrairement à la résistance, qui rend les antibiotiques totalement inefficaces, la tolérance et la persistance ne permettent donc pas aux bactéries de proliférer lorsqu’elles sont exposées à un antibiotique. En revanche, elles favorisent la survie des bactéries durant le traitement, permettant ainsi à celles encore vivantes lors de son arrêt de ré-établir une nouvelle population. L’un ou l’autre de ces phénomènes pourrait-il contribuer à la récurrence de certaines infections ? Pour répondre à cette question, il est essentiel de savoir reconnaître et distinguer ces deux situations. Pour cela, un test classique consiste à réaliser des courbes de survie (Figure 1), plus sombrement dénommées killing curves en anglais. Le principe est simple : on cultive les bactéries d’intérêt dans un milieu de culture (sorte de soupe nutritive pour bactéries), et on compte le nombre de bactéries encore vivantes au cours du temps après l’ajout d’un antibiotique. Les bactéries tolérantes meurent moins vite que les bactéries susceptibles, ce qui se traduit par une courbe de survie à pente plus faible (Figure 1, courbe jaune) que celle de ces dernières (Figure 1, courbe rose). Les bactéries persistantes, elles, présentent une courbe biphasique caractéristique, avec la majorité de la population tuée par l’antibiotique (courbe qui suit celle des bactéries susceptibles), à l’exception d’une minorité de bactéries persistantes (Figure 1, brusque aplatissement de la pente, courbe bleue).
Si les infections récurrentes sont bien documentées en clinique, l’investigation se limite souvent à distinguer les ré-infections (qui nécessitent une nouvelle rencontre avec la bactérie) des rechutes (la bactérie responsable de la première infection cause également la suivante). Le phénotype des bactéries isolées lors de ces rechutes est rarement caractérisé au laboratoire.
C’est cette lacune que l’équipe de la docteure Helaine propose de combler, afin de rendre les observations faites in vitro plus pertinentes dans un contexte clinique. Après tout, il faut connaître son ennemi pour pouvoir le vaincre. Et ce n’est pas César qui dira le contraire.
D’irréductibles salmonelles
L’ennemi sur lequel se concentre cette étude est la bactérie Salmonella enterica sous-espèce enterica sérotype Typhimurium, principale responsable de la salmonellose. Cette infection du tube digestif entraîne le plus souvent une gastro-entérite qui peut également devenir invasive en s’étendant à d’autres organes. Chez les patients immunodéprimés, les salmonelloses invasives ont tendance à récidiver après traitement [3, 4].
Un phénotype tolérant ou persistant pourrait-il être à l’origine de ces salmonelloses invasives récurrentes ? Pour le découvrir, Peter Hill et ses collègues déterminent le phénotype de salmonelles isolées chez 4 patients différents dont l’infection a rechuté une ou plusieurs fois [**]. Afin de reproduire au mieux les conditions auxquelles sont soumises ces bactéries lors d’une infection (nutriments limités, système immunitaire en alerte), les courbes de survie sont réalisées non pas en milieu de culture, mais au sein de macrophages. En effet, les macrophages sont des cellules immunitaires présentes dans tous les tissus et notamment au niveau de la muqueuse intestinale, cible privilégiée des salmonelles.
En temps normal, les macrophages assurent la défense de la muqueuse en phagocytant les bactéries qui se trouvent dans leur périmètre de surveillance. Les bactéries phagocytées sont alors détruites, et les macrophages alertent le reste du système immunitaire de la détection d’un intrus. Lors de certaines infections, les macrophages échouent dans leur rôle de défense et deviennent au contraire des réservoirs. Les bactéries phagocytées échappent alors à la digestion par les macrophages et les utilisent comme abri pour proliférer tout en éludant la réponse immunitaire. Une fois cette dernière calmée, les bactéries sont alors capables de ré-initier une infection [5].
Dans le cadre d’infections à salmonelles invasives récurrentes, les macrophages justement sont soupçonnés d’abriter des bactéries dissidentes responsables des rechutes [6]. C’est pourquoi l’équipe de recherche veut évaluer l’efficacité des antibiotiques contre ces bactéries dissimulées dans les macrophages, en adaptant le test de courbe de survie comme suit. Les échantillons cliniques de patients sont utilisés pour infecter des macrophages dérivés de souris grâce à 30 minutes d’incubation qui permettent aux bactéries d’être internalisées par phagocytose. Après cette incubation, un antibiotique, la céfotaxime, est ajouté pour une durée de 24 heures. À intervalles réguliers, une partie des macrophages est prélevée afin d’estimer le nombre de bactéries vivantes qu’ils contiennent. Pour ce faire, ces macrophages sont lysés et leur contenu est étalé sur des boîtes de Petri qui permettent la croissance des bactéries. Le nombre de bactéries vivantes (qui poussent sur la boîte) est alors compté.
La courbe de survie biphasique observée (Figure 1, courbe bleue) pour les quatre échantillons cliniques testés est sans appel : d’irréductibles salmonelles persistent encore et toujours face à l’antibiotique.
Recréer la potion magique en laboratoire
Ces données corroborent les observations faites sur plusieurs modèles d’infection à salmonelles Typhimurium précédemment développés au laboratoire. En effet, l’infection in vivo de souris ou in vitro de macrophages dérivés de ces souris a montré que les bactéries capables de survivre au traitement antibiotique présentaient un phénotype persistant (Figure 2).
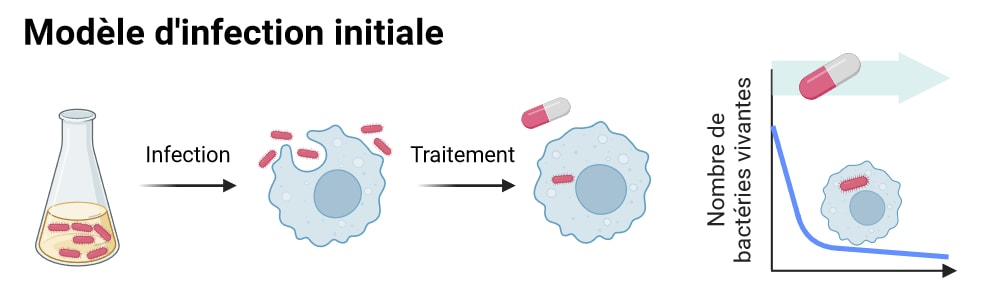
Cependant, ces modèles ne représentent en réalité que l’infection initiale, avant la ou les rechutes. Afin d’étudier les bactéries persistantes dans des conditions aussi proches que possible de celles d’une infection clinique récurrente, l’équipe de recherche décide de pousser le modèle plus loin, en y intégrant plusieurs cycles de traitement antibiotique.
Pour cela, le même modèle d’infection est simplement répété plusieurs fois : les bactéries sont mises en culture avant d’infecter les macrophages, ceux-ci sont ensuite traités à la céfotaxime et enfin lysés avant de récupérer les bactéries survivantes pour les remettre en culture et recommencer le cycle (Figure 3). L’alternance rechute/traitement est ainsi mimée.
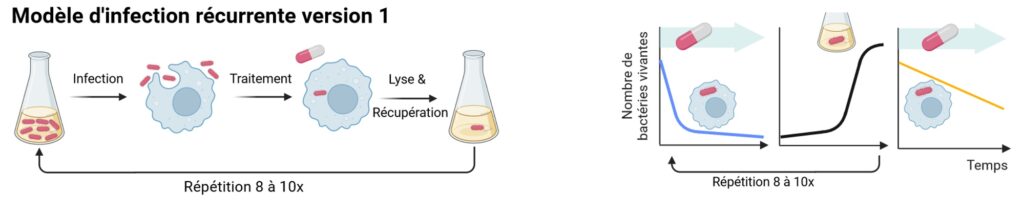
Les bactéries initiales présentent un phénotype persistant (Figure 3, schéma de droite, courbe bleue). Au bout de trois cycles de ré-infection/traitement, les chercheurs remarquent que le nombre de bactéries qui survit au traitement antibiotique augmente. Au bout de 10 cycles, le taux de survie peut même être jusqu’à 500 fois supérieur à celui des bactéries initiales ! Les chercheurs répètent l’expérience plusieurs fois pour confirmer cette observation : au bout de quelques cycles, les bactéries ont toujours un taux de survie supérieur à celui des bactéries initiales.
Le génome des populations bactériennes obtenues au bout de 10 cycles de traitement est analysé, afin de voir si cette augmentation du taux de survie peut s’expliquer par une sélection génétique, peut-être de traits favorisant la persistance. Dans cette étude, une mutation est considérée comme sélectionnée, et donc potentiellement bénéfique au taux de survie, si elle est présente dans au moins 50 % des bactéries séquencées à la fin d’une des expériences évolutives. 17 mutations codantes (c’est-à-dire situées dans un gène) sont ainsi identifiées, la plupart (15 sur 17) affectant des gènes impliqués dans le métabolisme.
Étonnamment, aucune mutation identifiée n’est commune avec les mutations observées dans les souches cliniques testées initialement. Les chercheurs veulent donc vérifier si ces mutations sélectionnées in vitro sont bien responsables de l’augmentation du taux de survie observé. Pour ce faire, ils réalisent des mutants de délétion de ces gènes [***]. Étant donné la surreprésentation de gènes liés au métabolisme dans le panel des mutations identifiées (15 sur les 17 identifiés), les efforts de l’équipe de recherche se concentrent sur ces gènes. La délétion de 6 de ces 15 gènes conduit effectivement à une augmentation significative du taux de survie des bactéries au sein des macrophages après traitement antibiotique par comparaison avec les bactéries initiales.
Le retrait du gène purA en particulier (impliqué dans la synthèse de la purine, qui compose notamment les acides nucléiques comme l’ADN) semble particulièrement bénéfique à la survie des bactéries dans des macrophages traités aux antibiotiques. Une courbe de survie est réalisée, afin de voir si le retrait de ce gène augmente le phénotype de persistance, ce qui expliquerait ainsi le meilleur taux de survie. Et là… surprise, les bactéries présentent un phénotype tolérant, avec une courbe de survie linéaire (Figure 3, schéma de droite, courbe jaune). Y aurait-il anguille sous menhir ?
En effet, les souches isolées chez des patients souffrant de salmonellose récurrente présentaient un phénotype persistant. Bien que la différence de phénotype observée in vitro par rapport à l’étude clinique puisse s’expliquer par plusieurs facteurs, notamment par le nombre plus élevé de cycles de traitement réalisé en laboratoire, les chercheurs questionnent la capacité des mutants métaboliques, pourtant sélectionnés par leur modèle, à ré-établir une infection. En effet, une étude précédente a établi qu’une souche de salmonelle (différente de celle utilisée dans l’étude), délétée de purA, était moins virulente chez des modèles murins, bien que capable de persister longtemps dans les tissus infectés [7]. Cela remet en cause la validité du modèle d’infection récurrente développé dans cette étude.
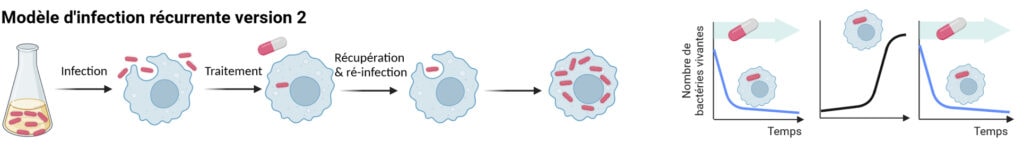
Le modèle est donc à nouveau adapté par l’équipe de recherche pour le rendre plus représentatif : au lieu de permettre aux bactéries survivantes de proliférer en milieu de culture, où les nutriments sont abondants, celles-ci sont directement incubées avec de nouveaux macrophages, pour juger de leur capacité à ré-établir une infection dans des conditions moins favorables (Figure 4). Et là, bingo ! Tandis que la souche bactérienne initiale, persistante (Figure 4, schéma de droite, graphe de gauche), est capable de recommencer à proliférer dans le macrophage (Figure 4, schéma de droite, graphe central), le mutant de délétion purA, tolérant, en est incapable. On dirait que le modèle mis au point par l’équipe tient enfin le bon bout : comme les souches récurrentes observées en clinique, les bactéries capables de ré-initier une infection in vitro ont un phénotype persistant en présence d’antibiotiques.
Une stratégie à haut risque
Les chercheurs peuvent enfin mettre leur modèle à profit pour tenter de comprendre les mécanismes de survie mis en place par les bactéries persistantes lors du traitement antibiotique des macrophages. Pour ce faire, ils décident de comparer les mutants tolérants et les bactéries persistantes, afin d’identifier des facteurs propres au maintien de la survie de ces dernières.
L’ARN des bactéries persistantes et tolérantes est séquencé en situation d’infection de macrophages traités à la céfotaxime, afin d’identifier les gènes différentiellement exprimés entre les deux phénotypes. Il en ressort que les gènes appartenant à la réponse SOS sont particulièrement surexprimés chez les bactéries persistantes par rapport aux bactéries tolérantes. La réponse SOS, qui porte bien son nom, tire la sonnette d’alarme chez les bactéries lorsque l’ADN est endommagé et présente des cassures double brin. Elle entraîne l’expression de plusieurs gènes, notamment impliqués dans la réparation des lésions de l’ADN.
Sous le microscope, l’équipe de recherche s’aperçoit que les bactéries persistantes présentent effectivement des cassures double brin tandis que les bactéries tolérantes ne semblent pas souffrir de dommages à l’ADN [****]. En creusant encore, un marquage Hoechst, qui colore l’ADN, révèle que les bactéries persistantes contiennent bien plus d’ADN que les bactéries tolérantes. Cela indique que, bien qu’elles ne se divisent pas en présence d’antibiotiques, les bactéries persistantes continuent de répliquer leur ADN. Les bactéries tolérantes, elles, sont fidèles à leur réputation de marmotte et ont mis toute activité de synthèse en arrêt (Figure 5, panel de gauche).
L’activité de néo-synthèse d’ADN chez les bactéries persistantes les expose aux mécanismes de défense des macrophages. Ceux-ci produisent notamment des molécules oxydantes qui réagissent avec les nucléotides synthétisés et incorporés dans le génome. La bactérie tente alors de retirer ces nucléotides abîmés de son génome, ce qui peut contribuer à générer des cassures double brin. Cette attaque immunitaire explique donc que le système de réparation SOS des bactéries persistantes tourne à plein régime pour réparer les dégâts (Figure 5, panel central).
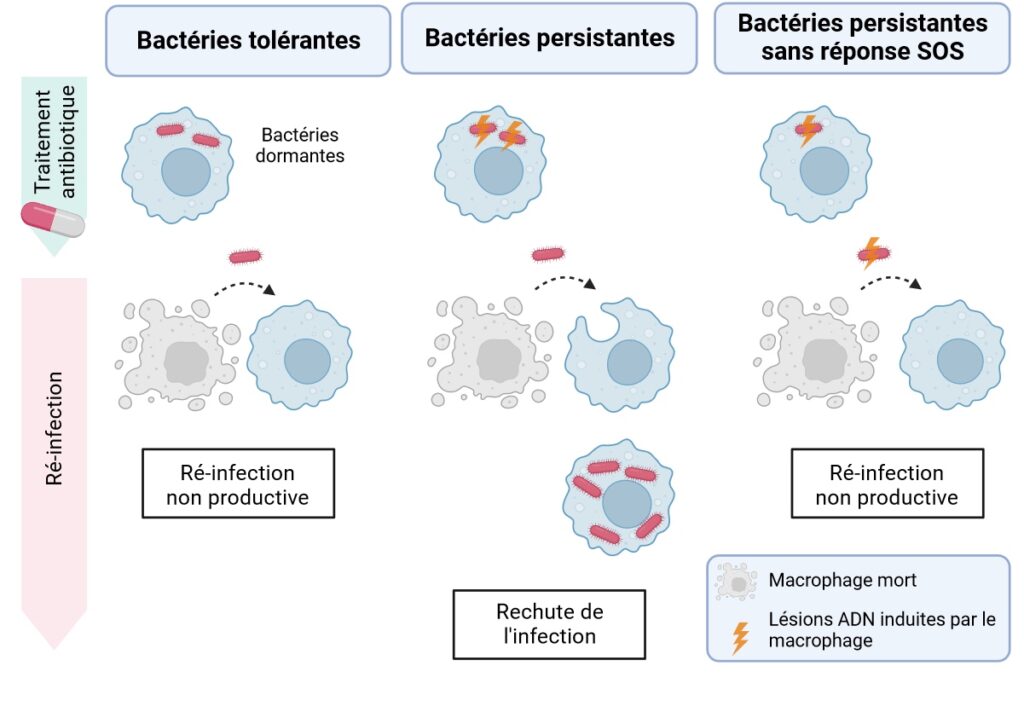
Finalement, pour les bactéries, on dirait que le maintien d’une activité métabolique, quitte à s’exposer aux défenses immunitaires, est indispensable pour survivre au sein des macrophages et garder la possibilité de ré-initier une infection. Comme joliment dit par les auteurs de la publication, « c’est la vulnérabilité des bactéries persistantes qui fait aussi leur force. »
Arrêtez ce Druide, par Neptune !
L’équipe de recherche montre en effet que, sans le gène de réparation des cassures double brin (recA) induit par la réponse SOS, le taux de survie après traitement antibiotique ainsi que la capacité des bactéries survivantes à proliférer à nouveau en milieu de culture sont toutes les deux sévèrement impactées (Figure 5, panel de droite). Pas complètement éliminées cependant, ce qui suggère l’importance d’autres mécanismes, mais c’est un bon début !
Dans un premier essai in vivo, des souris sont infectées par des salmonelles délétées de recA. À la fin du traitement antibiotique, il reste beaucoup moins de bactéries vivantes dans les tissus que lorsqu’on infecte les souris avec des bactéries non modifiées. Plus prometteur encore, il n’y a eu aucune rechute de l’infection chez les souris infectées avec les bactéries sans système de réparation, au contraire des bactéries sauvages. Le blocage de la réponse SOS serait-il la clé pour permettre aux antibiotiques de venir à bout des salmonelloses qui rechutent ? Espérons une réponse dans les prochains tomes.
[*] Alexander Fleming a découvert la pénicilline en 1928 et publié ses observations en 1929 [8]. Pour en savoir plus sur l’histoire des antibiotiques, vous pouvez également consulter cet article [9], ou écouter l’épisode 50 du podcast “This Podcast Will Kill You” (en anglais).
[**] Ces échantillons ont été collectés lors d’une étude clinique précédente, réalisée par une équipe indépendante [1].
[***] Un mutant de délétion est dit d’un organisme (ici, une bactérie) auquel on a retiré un gène d’intérêt via des techniques de biologie moléculaire. Dans cette publication, le retrait des gènes mutés permet de détecter les gènes dont l’inactivité (induit par la mutation) augmente le taux de survie.
[****] Les cassures sont notamment observées via un essai TUNEL (terminal deoxynucleotidyl transferase (TdT) mediated dUTP nick-end labeling). Cette analyse consiste à marquer les cassures en exploitant l’activité d’une enzyme (appelée transférase terminale de deoxynucleotidyl) qui catalyse l’addition de dUTP, ici modifiés pour être fluorescents, aux extrémités 3’ libres de l’ADN. Les bactéries ainsi marquées peuvent alors être observées au microscope.
[1] Abraham E.P. & Chain E., An Enzyme from Bacteria able to Destroy Penicillin. Nature, 1940. DOI : 10.1038/146837a0. [Publication scientifique]
[2] Fisher R.A., et al., Persistent bacterial infections and persister cells. Nature Reviews Microbiology, 2017. DOI : 10.1038/nrmicro.2017.42. [Review]
[3] Organisation Mondiale de la Santé, Salmonella (infections à, non typhiques). OMS, 2018. [Site internet, consulté le 13/05/2023]
[4] Okoro C.K., et al., High-resolution single nucleotide polymorphism analysis distinguishes recrudescence and reinfection in recurrent invasive nontyphoidal Salmonella typhimurium disease. Clinical Infectious Diseases: An Official Publication of the Infectious Diseases Society of America, 2012. DOI : 10.1093/cid/cir1032. [Publication scientifique]
[5] Price J.V. & Vance R.E., The macrophage paradox. Immunity, 2014. DOI : 10.1016/j.immuni.2014.10.015. [Review]
[6] Gordon M.A., et al., Invasive Non-typhoid Salmonellae Establish Systemic Intracellular Infection in HIV-Infected Adults: An Emerging Disease Pathogenesis. Clinical Infectious Diseases, 2010. DOI : 10.1086/651080. [Publication scientifique]
[7] O’Callaghan D. et al., Characterization of aromatic- and purine-dependent Salmonella typhimurium: attention, persistence, and ability to induce protective immunity in BALB/c mice. Infection and Immunity, 1988. DOI : 10.1128/iai.56.2.419-423.1988. [Publication scientifique]
[8] Fleming A., On the Antibacterial Action of Cultures of a Penicillium, with Special Reference to Their Use in the Isolation of B. Influenzæ. British Journal of Experimental Pathology, 1929. PMCID : PMC2048009. [Publication scientifique]
[9] Gould K., Antibiotics: from prehistory to the present day. Journal of Antimicrobial Chemotherapy, 2016. DOI : 10.1093/jac/dkv484. [Review]