Écriture : Matthieu Nicault
Relecture scientifique : Élodie Billard
Relecture de forme : Maud Billaud et Dorian Vigoureux
Temps de lecture : environ 12 minutes.
Thématiques : Microbiologie et Génétique & Épigénétique (Biologie)
Publication originale : Schroeckh V., et al., Intimate bacterial-fungal interaction triggers biosynthesis of archetypal polyketides in Aspergillus nidulans. Proceedings of the National Academy of Sciences, 2009. DOI : 10.1073/pnas.0901870106

Dans leur article de 2009, Schroeck et son équipe se sont intéressés aux interactions entre des microorganismes, plus particulièrement entre une bactérie du genre Streptomyces et un champignon Aspergillus nidulans. Ils ont d’abord démontré que le mécanisme de l’interaction repose sur un contact étroit entre les deux organismes. Ensuite, ils ont observé que cette interaction conduit à l’activation de certains gènes et à la modification de leur expression ; menant à la production de nouvelles molécules antimicrobiennes.
Les microorganismes sont présents dans tous les milieux et vivent le plus souvent en communautés. Au sein de celles-ci, ils communiquent entre eux en utilisant des molécules spécialisées appelées métabolites. Dans un article paru en 2009, des chercheurs ont étudié le mécanisme des interactions entre des bactéries et un champignon pour comprendre comment elles pouvaient se dérouler. Pour cela, ils ont réalisé des co-cultures entre 58 bactéries et un champignon, c’est-à-dire qu’ils ont fait pousser une espèce de bactérie et le champignon sur un même milieu nutritif, en parallèle pour 58 espèces de bactéries différentes. Sur ces 58 modèles testés, ils sont parvenus à montrer que l’interaction entre une bactérie et un champignon parvient à augmenter l’expression de certains gènes codant pour la production, chez ce dernier, de métabolites spécialisés. Ensuite, ils sont parvenus à isoler quatre molécules connues pour tuer des microbes : des antimicrobiens. Les interactions bactérie/champignon jouent également un rôle écologique très important dans les écosystèmes et les molécules qu’elles induisent pourraient devenir essentielles pour la santé humaine.
Les microorganismes : une source de molécules au potentiel inexploité
Les microorganismes comme les bactéries et certains champignons sont retrouvés dans de nombreux écosystèmes tels que le sol ou les écosystèmes marins. Il y en a même dans le microbiote de certains organismes, comme par exemple dans notre intestin [1]. Ces microorganismes contribuent grandement à l’équilibre des écosystèmes auxquels ils appartiennent en participant à la dégradation de la matière organique dans les sols ou en produisant des molécules permettant de lutter contre d’autres microorganismes pathogènes par exemple [2].
D’ailleurs, de nombreuses molécules utiles à l’espèce humaine sont produites par des microorganismes. En effet, après les plantes, ce sont les principaux producteurs d’antibiotiques [3]. Les bactéries du genre Streptomyces et certains champignons microscopiques du genre Aspergillus produisent plus de 10 000 composés utiles pour nous, dont plus de la moitié sont des antibiotiques (Figure 1) [3]. Néanmoins, certaines molécules produites par les microorganismes ne possèdent pas d’activité antimicrobienne contre des microorganismes potentiellement dangereux pour l’être humain, les animaux ou les plantes tandis que d’autres molécules peuvent avoir des effets indésirables pour les cellules humaines. Ces molécules peuvent toutefois avoir un rôle important au sein de la communauté microbienne.
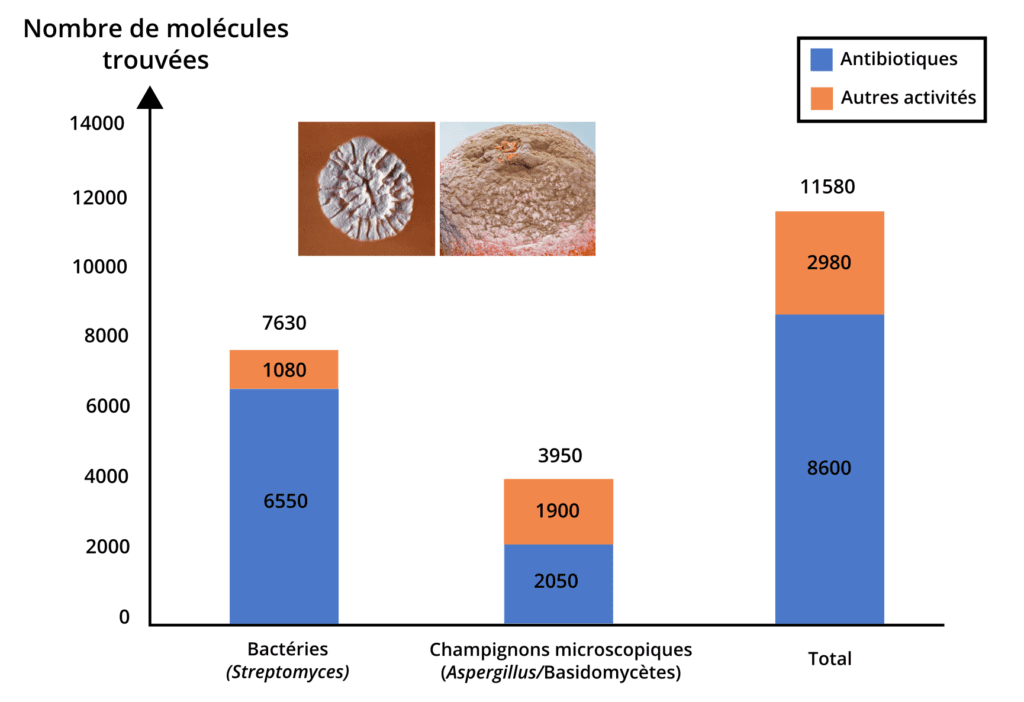
Souvent, ces microorganismes ne sont exploités que pour la production d’un seul composé. Par exemple, pendant longtemps la bactérie Streptomyces griseus a été utilisée essentiellement pour la production d’une molécule antibiotique : la streptomycine [4]. Cette molécule est utilisée contre la peste depuis 1947. Pourtant, l’analyse de l’ADN de souches de bactéries environnementales du genre Streptomyces a montré qu’en réalité, ces bactéries sont capables de produire entre 20 et 50 molécules d’intérêts [5]. Néanmoins, certaines molécules peuvent être produites à l’identique par plusieurs souches de bactéries différentes. Ainsi, avec seulement 8 souches différentes de bactéries du genre Streptomyces, le potentiel de production est de plus de 90 molécules uniques (sur 261 initialement détectées) [6]. De plus, ces molécules possèdent différentes natures chimiques et différents rôles pour les bactéries : elles appartiennent au métabolisme secondaire qui leur est utile pour se défendre ou s’adapter à un nouvel environnement. Par exemple, lorsqu’un microorganisme s’attaque à un autre ou à une colonie bactérienne, alors ces derniers se défendent en produisant des molécules qui éloignent ou tuent l’envahisseur. Ainsi, les bactéries ne synthétisent pas tout le temps ces molécules. Si l’on veut pouvoir tester et produire ces composés d’intérêt pour l’espèce humaine, il est donc primordial de trouver des méthodes pour le faire au bon moment. Une solution serait d’induire cette production en mimant l’envahissement d’une bactérie sur le terrain de jeu d’un champignon.
Les communautés microbiennes : le centre d’une discussion chimique
Durant des décennies, nous n’avons pas considéré l’importance des interactions entre les différents microorganismes. Mais en réalité, lorsqu’ils partagent le même habitat, voire un micro-habitat, ils possèdent la faculté d’interagir entre eux. Dans un gramme de sol, il peut co-exister plus d’un milliard de microorganismes divisés en des milliers d’espèces différentes [7]. Cette densité importante de population dans un endroit aussi restreint conduit à des interactions entre les microorganismes, ce qui implique la production de métabolites spécialisés [7]. Ces composés sont un moyen de communication pacifiste mais aussi une arme de défense. Les produits issus de ces interactions participent à l’équilibre des écosystèmes et peuvent aussi être utiles à l’espèce humaine, aussi bien au sein de son microbiote que par l’activité des molécules induites [7, 8, 9, 10].
C’est sur ce constat qu’une équipe de scientifiques a cherché à étudier ces interactions, en cultivant deux microorganismes ensemble, pour en comprendre les mécanismes et vérifier la production de métabolites spécialisés. Les deux microorganismes choisis sont une bactérie du genre Streptomyces, et un champignon appelé Aspergillus nidulans. Lors de leur co-culture, il est possible de voir au microscope le contact étroit existant entre la bactérie et le champignon.On observe que les deux organismes se développent sous la forme de filaments microscopiques, avec les bactéries de part et d’autre du champignon (Figure 2). Un contact s’établit donc entre les filaments des deux organismes.
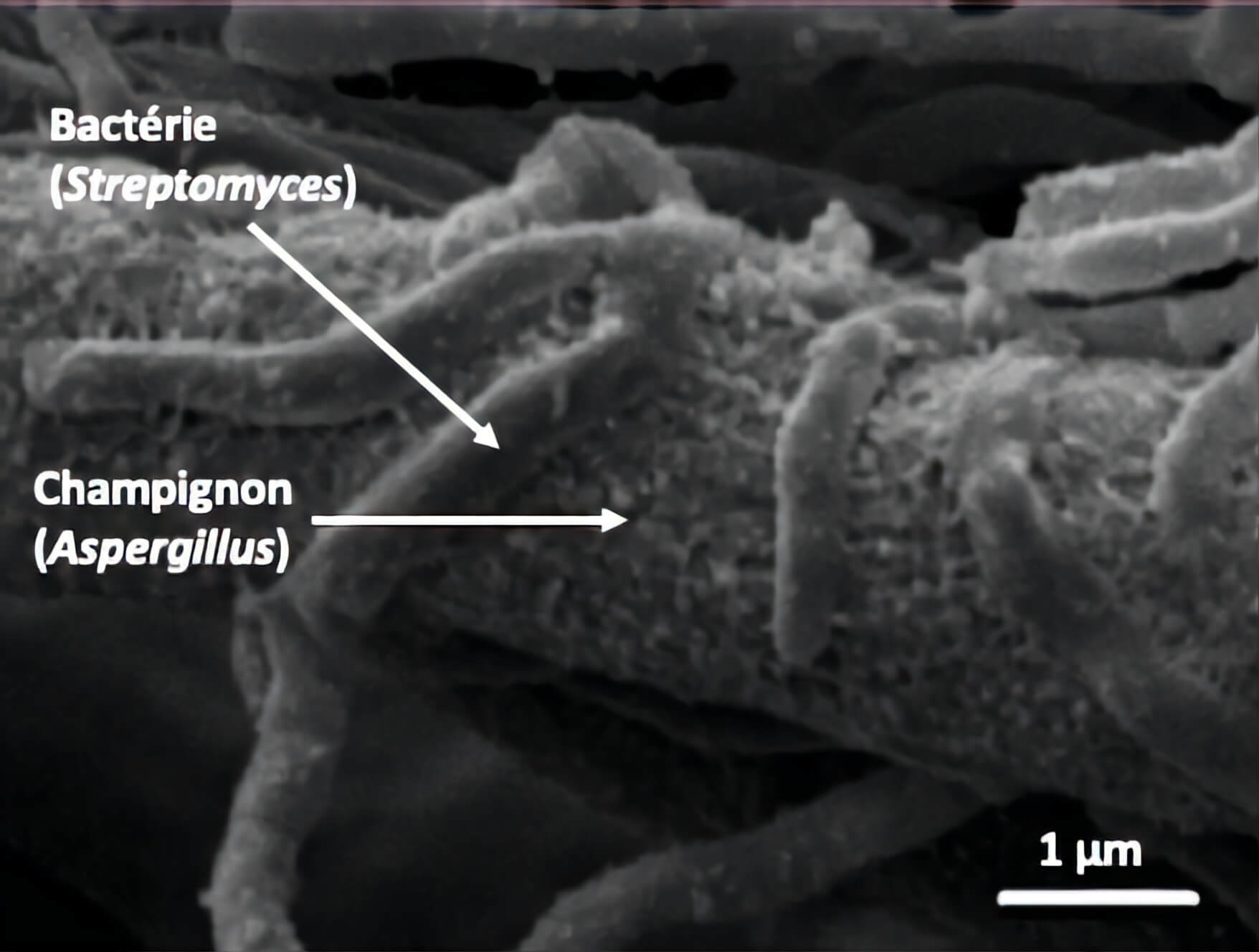
Les auteurs de la publication ont alors souhaité savoir si ce contact pouvait produire un signal induisant la production de métabolites, et si oui, de caractériser les changements dans le métabolisme du champignon, c’est-à-dire dans l’ensemble de ses processus biochimiques internes.
L’interaction microbienne : un phénomène qui se situe au niveau de l’expression des gènes…
Les métabolites microbiens spécialisés sont le fruit d’un ensemble de réactions chimiques appelées voies de biosynthèse, qui peuvent se produire grâce à certaines protéines : des enzymes. Pour répondre à leur question, les auteurs de la publication se sont alors intéressés à ces enzymes, et plus particulièrement à l’information génétique présente dans l’ADN qui permet de les former. Étant donné que ces enzymes ne sont pas utilisées en permanence par les microorganismes, leurs gènes ne sont donc activés — exprimés — qu’en cas de nécessité. Le reste du temps ils sont dit silencieux.
Dans la première partie de l’étude, les auteurs ont voulu voir si l’expression des gènes du champignon mis en contact avec la bactérie était modifiée. Or, l’expression d’un gène en protéine se fait en deux étapes : d’abord en passant de l’ADN à de l’ARN ; puis de l’ARN à une protéine. Pour cela, ils ont donc utilisé une méthode dite de qRT-PCR (pour « Réaction de polymérase en chaîne quantitative ») utilisée pour détecter l’ARN. Beaucoup d’entre vous ont dû entendre parler de la RT-PCR récemment car elle permet de détecter la présence de certains virus à ARN dans les fluides biologiques humains (sang, urine, mucus), comme le SARS-CoV-2 responsable de la CoViD-19. Ici, grâce à l’ajout d’un module de quantification, les auteurs de cet article ont été capables de mesurer précisément la différence de production d’ARN du champignon en présence ou en absence de la bactérie. Au cours de cette analyse, ils ont démontré que l’expression de près de 395 gènes est modifiée. 60 % de ces gènes, soit 248, sont exprimés davantage lorsque le champignon et la bactérie sont en contact, tandis que 40 % de ces gènes sont moins exprimés.
Grâce à la connaissance de l’intégralité de la séquence ADN de ce champignon, les chercheurs sont parvenus à identifier certains gènes codant justement pour des enzymes des voies de biosynthèse des métabolites spécialisés. Parmi les 248 gènes davantage exprimés, 5 proviennent d’une même voie de biosynthèse et participent à la formation de métabolites spécialisés (Figure 3, gènes 2 à 6). Ils ont inclus deux contrôles (négatif et positif) dans leur analyse afin de confirmer la fiabilité de la méthode et des résultats obtenus (Figure 3, gène 1 et gène 7, respectivement).
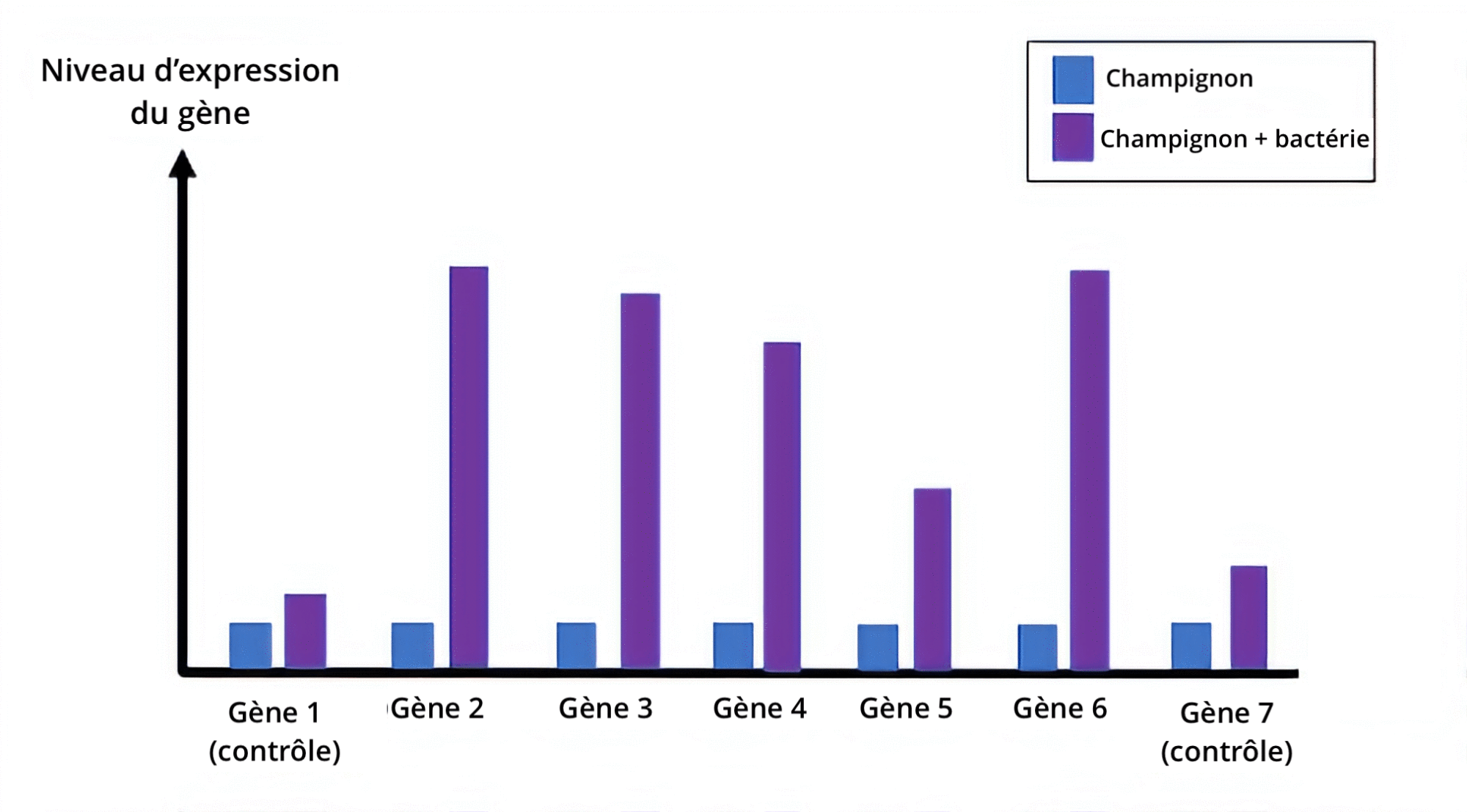
Ils en ont conclu que cette expression de gènes était la résultante du contact physique entre les deux microorganismes. Ils ont ensuite vérifié si l’activation de cette voie de biosynthèse induit bel et bien la production de nouveaux composés.
… et qui se retrouve au niveau de la production de molécules
Afin de confirmer la production de nouveaux métabolites spécialisés et plus précisément les produits de cette voie de biosynthèse, les auteurs ont effectué le profil chimique de cette interaction. Pour cela, il existe une méthode appelée chromatographie en phase liquide, qui permet de séparer les différentes molécules en fonction de paramètres physiques et chimiques et de déterminer leur nature. Pour image, rappelez-vous Abby dans NCIS qui utilise son spectromètre de masse et annoncer qu’ils ont retrouvés différents types de molécules dans le sang de la victime. Ici c’est pareil, les chercheurs ont pris le mélange de composés issu de l’interaction entre la bactérie et le champignon et l’ont comparé aux molécules produites par le champignon cultivé seul pour voir ce qui était différent. Cette analyse a mis en évidence la production de quatre composés dans la co-culture : sur les chromatogrammes obtenus, en Figure 4, on observe en effet quatre pics correspondant à une variation du signal liée à la présence de quatre molécules apparaissant spécifiquement sur le tracé en présence de la bactérie. Ces quatre pics ne sont pas présents pour le champignon seul.
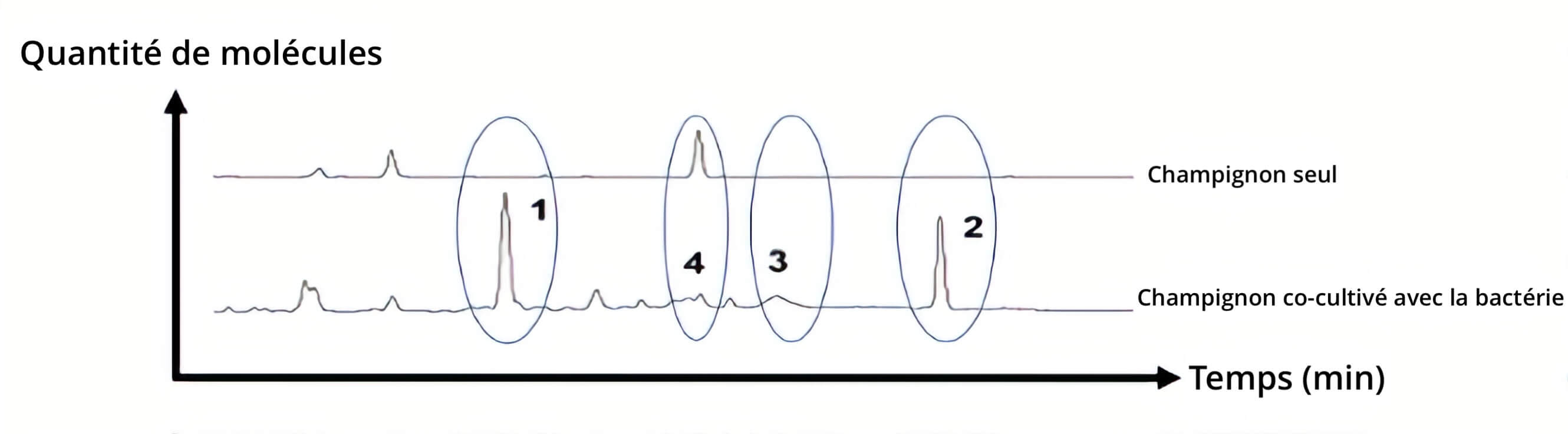
Ces composés possèdent des activités antimicrobiennes, évaluées par des tests où ils sont mis en présence de différents types de bactéries plus ou moins pathogènes pour nous, comme par exemple Staphylococcus aureus ou Escherichia coli. On observe alors que les bactéries sont tuées. Ces molécules au large spectre d’action seraient intéressantes dans le cadre de la lutte contre des bactéries multirésistantes aux antibiotiques déjà commercialisés.
Une source riche, essentielle et urgente à exploiter
De plus en plus d’études de ce tenant sont réalisées afin de trouver de nouvelles molécules antimicrobiennes [11]. En effet, une menace encore plus importante que la CoViD-19 pèse sur l’humanité : les bactéries multirésistantes aux antibiotiques. L’utilisation abusive des antibiotiques, que ce soit pour la santé humaine ou pour l’élevage animal, a conduit à l’émergence de bactéries capables de résister à ces molécules. Actuellement, de nombreux décès liés à ces bactéries multirésistantes sont recensés, avec près de 30 000 décès en Europe et 700 000 dans le monde chaque année [6]. Ce phénomène pourrait avoir des conséquences dramatiques puisqu’en 2050, il y aura plus de morts dues à des maladies d’origine bactérienne qu’à la seule maladie du cancer (10 000 000 pour les microorganismes multi-résistants contre 8 000 000 pour les cancers [12]).
Il est donc important de prendre soin de son environnement, car il pourrait nous sauver la vie de bien des façons. Par exemple, les interactions microbiennes décrites dans cet article conduisent parfois à la redécouverte de molécules déjà connues, mais il existe aussi des méthodes qui permettent de ne détecter que des composés inconnus. De plus, il existe d’autres méthodes permettant d’exploiter toute la diversité microbienne présente dans le sol, sans se limiter aux 1 % des microorganismes qui peuvent être cultivés en laboratoire.
Ces nouvelles méthodes de prospection que sont les co-cultures microbiennes devraient permettre de lever le voile sur des molécules jusque-là non caractérisées.
Éléments pour approfondir
Dans cet article, les auteurs ont étudié un type d’interaction entre microorganismes : un contact cellulaire direct entre une bactérie et un champignon microscopique. L’étude des interactions entre microorganismes est une discipline de la microbiologie appelée socio-microbiologie. Ce terme est apparu en 2005 quand les deux chercheurs Matt Parsek et Peter Greenberg ont décrit les comportements des microorganismes en communauté [13]. Contrairement aux humains, ces contacts ne passent pas que par un contact physique comme une poignée de main, se faire la bise ou se battre en cas de conflit, mais par d’intenses échanges de molécules. Parmi les mécanismes les plus connus et les plus caractérisés au niveau des interactions microbiennes et de la formation de ces molécules, on trouve par exemple :
- le quorum-sensing ;
- la formation de biofilm ;
- le phénomène d’antibiose [14].
Le quorum-sensing est un phénomène auto-induit en lien avec la densité de population au sein d’une communauté microbienne. Ce système de communication induit une modification de l’expression de gènes spécifiques pour certaines fonctions essentielles, ainsi que la production de molécules qui permettent l’adaptation de la communauté et de sa croissance à son milieu de vie. Pour résumer le quorum-sensing, des molécules sont produites et indiquent à tout le monde « mettons-nous en marche ensemble et dans la même direction ».
Quant à la formation de biofilms, elle résulte de la réponse à des paramètres environnementaux (présence de stress biologiques et/ou physico-chimiques, niche écologique favorable, vie en communauté) sur une communauté bactérienne. Lorsqu’un signal environnemental est perçu ou en présence de molécules de quorum-sensing, les bactéries vont former un agrégat polysaccharidique appelé biofilm. Autrement dit, c’est le principe de « l’union fait la force ».
La dernière approche est celle de l’antibiose. C’est une interaction très connue qui se déroule lorsqu’une espèce est soumise à une pression biotique : présence d’un autre organisme ou de substances métaboliques produites par un autre organisme. Ces relations dites antagonistes vont parfois conduire les bactéries à produire des signaux qui empêchent l’envahisseur d’atteindre la colonie. Là c’est plutôt « attention tu es sur mon territoire si tu t’approches trop ça va barder ! », allant parfois jusqu’au « bacteriocide » (par analogie avec l’homicide).
Toutes ces interactions reposent sur la production de métabolites spécialisés. Certaines de ces molécules possèdent une activité antimicrobienne et peuvent être utilisées par l’espèce humaine.
À cela s’ajoute un autre domaine de recherche intéressant pour l’avenir : les métabolites spécialisés comme molécules de signalisation. Il existe une diversité importante de molécules de signalisation dont certaines permettent de produire les métabolites spécialisés ayant des effets antibiotiques. Dans le cas des antibiotiques, ces molécules de signalisation peuvent avoir différentes conséquences sur l’organisme récepteur. D’un côté, l’antibiotique à forte dose peut entraîner la mort cellulaire, mais d’un autre côté, à faible dose, il agit comme une molécule de signalisation et entraîne à son tour la production d’autres composés. Par exemple, chez une espèce de bactéries du genre Streptomyces, la présence de concentrations différentes de deux antibiotiques a conduit à des réponses physiologiques et métaboliques différentes et notamment à la production de composés antimicrobiens [15]. L’utilisation d’antibiotiques déjà connus, à de faibles quantités, dans le milieu où les bactéries sont cultivés pourrait, de part leur rôle de molécule signal, induire la production de nouvelles molécules.
[1] Tourneroche A., et al., Bacterial–Fungal Interactions in the Kelp Endomicrobiota Drive Autoinducer-2 Quorum Sensing. Frontiers in Microbiology, 2019. DOI : 10.3389/fmicb.2019.01693. [Publication scientifique]
[2] Rousk J. & Bengtson P., Microbial regulation of global biogeochemical cycles. Frontiers in Microbiology, 2014. DOI : 10.3389/fmicb.2014.00103. [Publication scientifique]
[3] Bérdy J., Bioactive Microbial Metabolites: A Personal View. The Journal of Antibiotics, 2005. DOI : 10.1038/ja.2005.1. [Publication scientifique]
[4] Garson W.& Waksman S. A., Strain Specificity and Production of Antibiotic Substances: VII. Production of a Grisein-Like Antibiotic by a Strain of Streptomyces Griseus. PNAS, 1948. DOI : 10.1073/pnas.34.6.232. [Publication scientifique]
[5] Baltz, R.H., Gifted microbes for genome mining and natural product discovery. J Ind Microbiol Biotechnol, 2017. DOI : 10.1007/s10295-016-1815-x. [Publication scientifique]
[6] Nicault M., et al., Mining the Biosynthetic Potential for Specialized Metabolism of a Streptomyces Soil Community. Antibiotics, 2020. DOI : 10.3390/antibiotics9050271. [Publication scientifique]
[7] Traxler M. F. & Kolter R., Natural products in soil microbe interactions and evolution. Natural Product Reports, 2015. DOI : 10.1039/C5NP00013K. [Publication scientifique]
[8] Deveau A., et al., Bacterial–fungal interactions: ecology, mechanisms and challenges. FEMS Microbiology Reviews, 2018. DOI : 10.1093/femsre/fuy008. [Publication scientifique]
[9] Diaz P. I., et al., Fungal-bacterial interactions and their relevance to oral health: linking the clinic and the bench. Frontiers in Cellular and Infection Microbiology, 2014. DOI : 10.3389/fcimb.2014.00101. [Publication scientifique]
[10] Frey-Klett P., et al., Bacterial-Fungal Interactions: Hyphens between Agricultural, Clinical, Environmental, and Food Microbiologists. Microbiology and Molecular Biology Reviews, 2011. DOI : 10.1128/MMBR.00020-11. [Publication scientifique]
[11] Bertrand S., et al., Metabolite induction via microorganism co-culture: A potential way to enhance chemical diversity for drug discovery. Biotechnology Advances, 2014. DOI : 10.1016/j.biotechadv.2014.03.001. [Publication scientifique]
[12] Rapport final de la Revue de la résistance aux antimicrobiens (The Review on Antimicrobial Resistance) disponible en anglais ici : https://amr-review.org/
[13] Parsek M. R. & Greenberg E. P., Sociomicrobiology: the connections between quorum sensing and biofilms. Trends in Microbiology, 2005. DOI : 10.1016/j.tim.2004.11.007. [Publication scientifique]
[14] Cossart P., « La nouvelle microbiologie – Des microbiotes aux CRISPR », Éditions Odile Jacob Sciences, Paris, 2016. [Livre de science]
[15] Romero D., et al., Antibiotics as Signal Molecules. Chemical Reviews, 2011. DOI : 10.1021/cr2000509. [Publication scientifique]