Ce papier-mâché a été écrit par des étudiantes et étudiants de Master 1 du Magistère Européen de Génétique de l’Université Paris-Cité, dans le cadre d’une collaboration pédagogique pour l’UE Communication scientifique.
Promotion 2024-2025
Écriture : Adrien Hallais, Taïs Victor Bergevin, Gabriel Karrillon, Eva Lebel Bélinguier
Relecture scientifique : Équipe pédagogique : Sandra Claret et Patrick Laurenti
Relecture de forme : Eléonore Pérès
Temps de lecture : environ 10 minutes.
Thématiques : Génétique, Développement (Biologie)
Publication originale : Westerich K. J., et al., Spatial organization and function of RNA molecules within phase-separated condensates in zebrafish are controlled by Dnd1. Developmental cell, 2023. DOI : 10.1016/j.devcel.2023.06.009

Les biocondensats, des structures sans membrane dans les cellules, sont au cœur de la régulation de la traduction des ARNm, un processus essentiel pour la différenciation des cellules germinales. Cette étude explore un mécanisme spécifique de cette régulation, montrant comment des acteurs moléculaires comme Dnd1 et nanos3 jouent un rôle clé dans la détermination du destin des précurseurs germinaux et influencent leur développement.
Dans notre corps, les cellules se répartissent en deux groupes : les cellules somatiques, qui constituent la majorité de nos organes et tissus et les cellules germinales qui sont spécifiques à la reproduction. Les cellules germinales sont cruciales pour la transmission du patrimoine génétique à la prochaine génération. Le processus de leur développement et de leur différenciation est au cœur de ce papier-mâché.
Tout commence avec la cellule œuf, une cellule totipotente, capable de se différencier en tous les types de cellules. À partir de cette cellule unique, les premières divisions cellulaires aboutissent à des cellules indifférenciées, sans fonction précise. Puis, ces cellules se spécialisent progressivement : certaines deviennent des cellules somatiques, tandis que d’autres se transforment en cellules germinales. Parmi toutes les cellules, les premières à acquérir une fonction sont les cellules germinales [1]. Ce processus de différenciation est essentiel pour la formation des gamètes (spermatozoïdes ou ovules) et pour assurer la continuité génétique.
Un acteur central dans cette régulation est la protéine Nanos3, présente chez tous les animaux. Nanos3 joue un rôle clé dans la transformation des cellules germinales indifférenciées en gamètes fonctionnels. Mais pour que cette protéine soit produite au bon moment et au bon endroit, une régulation précise est nécessaire. C’est là que les biocondensats interviennent : ces structures sans membrane, présentes dans les cellules germinales, jouent un rôle fondamental dans l’organisation et la régulation des ARN messagers (ARNm).
La protéine Dnd1, par exemple, est essentielle au bon fonctionnement des biocondensats. Elle régule la traduction des ARNm spécifiques comme celui de Nanos3 au sein de ces granules germinaux. Ces granules sont des compartiments cellulaires spécialisés qui stockent et protègent les ARNm, permettant ainsi de contrôler la production de protéines cruciales pour le développement des cellules germinales. Cette organisation est indispensable à la bonne différenciation des cellules germinales, une étape clé pour garantir la formation des gamètes et la transmission du matériel génétique.
Bien que les biocondensats aient été identifiés chez de nombreux organismes, tels que le poisson-zèbre, la drosophile et le nématode C. elegans, de nombreuses questions demeurent sur la manière dont ces structures se forment et régulent l’expression des gènes. Comment l’organisation des ARNm à l’intérieur de ces granules affecte-t-elle le destin des cellules germinales ? C’est ce genre de mécanisme de régulation que nous allons explorer dans ce papier-mâché.
Interaction : tout est une question de proximité
Mais par où commencer pour comprendre ces mécanismes complexes ? L’équipe de recherche de cette publication s’est d’abord concentrée sur la détermination du rôle de Dnd1 dans les granules germinaux et sur comment cette protéine interagit avec l’ARNm nanos3 au sein de ces structures.
Le meilleur moment pour observer la localisation des molécules dans les granules est 10 heures après la fertilisation, stade où les granules atteignent leur plus grande taille. Pour suivre précisément les groupements de Dnd1 (une protéine) et de nanos3 (un ARNm), les scientifiques ont utilisé différentes méthodes pour marquer et localiser ces groupements dans les cellules :
Le marquage des protéines : pour localiser les protéines, on utilise une méthode appelée immunomarquage. Cela consiste à utiliser un anticorps, une molécule qui se fixe spécifiquement à la protéine que l’on souhaite observer. Cette molécule a été précédemment reliée à une molécule fluorescente sous un type d’éclairage spécifique, appelée fluorochrome. Cela permet d’observer la protéine au microscope lorsqu’on l’illumine à la lumière ultraviolette.
Suivre l’ARNm avec le système PP7 : le système PP7 est une technique qui permet de repérer l’ARNm dans les cellules. Les chercheurs ont ajouté de petites étiquettes (appelées boucles de tige PP7) sur l’ARNm nanos3. Ces étiquettes permettent à un fluorochrome de s’y accrocher, ce qui permet de suivre en temps réel la localisation de cet ARNm dans les granules.
La microscopie confocale : cette technique permet de prendre des photos en « tranches » de l’intérieur des cellules, comme si l’on coupait un saucisson et photographiait précisément chaque couche. Grâce à ces images, les scientifiques peuvent reconstruire l’intérieur de la cellule en 3D et observer la proximité des différentes molécules. Si elles se trouvent à côté sur la même « tranche », cela signifie qu’elles sont en contact et peuvent interagir.
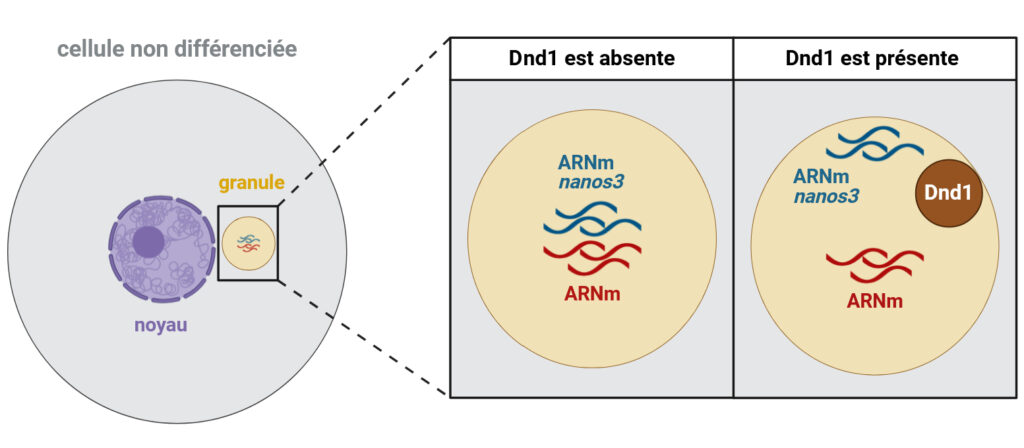
Grâce à ces techniques, l’équipe de recherche a montré que les ARNm nanos3 et la protéine Dnd1 se localisent et se superposent en périphérie des granules (Figure 1). Lorsque Dnd1 est retirée des granules dans une cellule non différenciée, les ARNm nanos3 restent au centre, au milieu d’autres ARNm (Figure 1). En présence de Dnd1 à la périphérie, celle-ci attire les ARNm nanos3 vers cette zone.
Cela a un impact majeur sur le fonctionnement de la cellule : lorsque les ARNm sont localisés au centre du granule, ils sont protégés de la dégradation et deviennent inaccessibles à d’autres protéines, à l’exception de celles présentes au centre du granule, ce qui rend leur traduction impossible. Cependant, à la périphérie des granules, l’ARNm devient plus accessible aux protéines présentes dans le cytoplasme, notamment aux ribosomes, les molécules qui traduisent l’ARNm en protéines.
Dnd1 : un acteur clé de la traduction des ARNm
L’interaction entre les groupements de Dnd1 et d’ARNm nanos3 joue-t-elle un rôle dans le fonctionnement des granules germinaux ?
Pour répondre à cette question, l’équipe de recherche a utilisé les techniques précédemment discutées, complétées par un immunomarquage spécifique permettant de localiser les ribosomes : un anticorps cible la grande sous-unité du ribosome (en violet, Figure 2) et un autre cible la petite sous-unité (en rose, Figure 2).
Les résultats de cette expérience ont confirmé la présence superposable des groupements d’ARNm nanos3, de Dnd1 et des ribosomes. Non seulement Dnd1 rapproche les deux composants nécessaires à la traduction de l’ARNm nanos3 (Figure 2), mais elle stabilise également leur interaction, tout en jouant un rôle de « protecteur ». En effet, des microARNs (comme mir-430) peuvent agir comme des « saboteurs » en bloquant l’association entre l’ARNm nanos3 et le ribosome, ce qui inhibe la traduction.
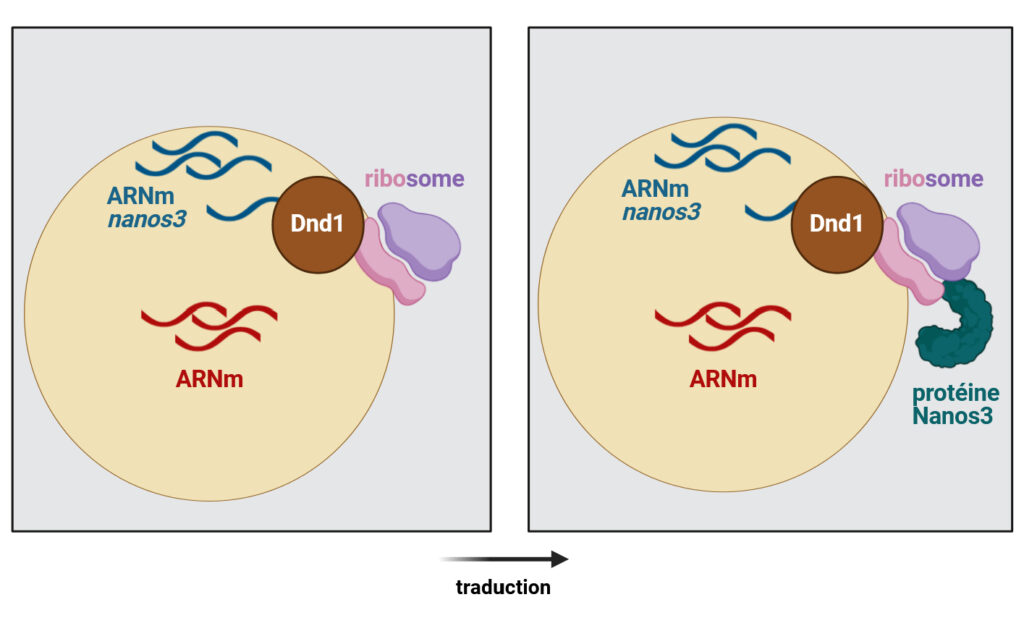
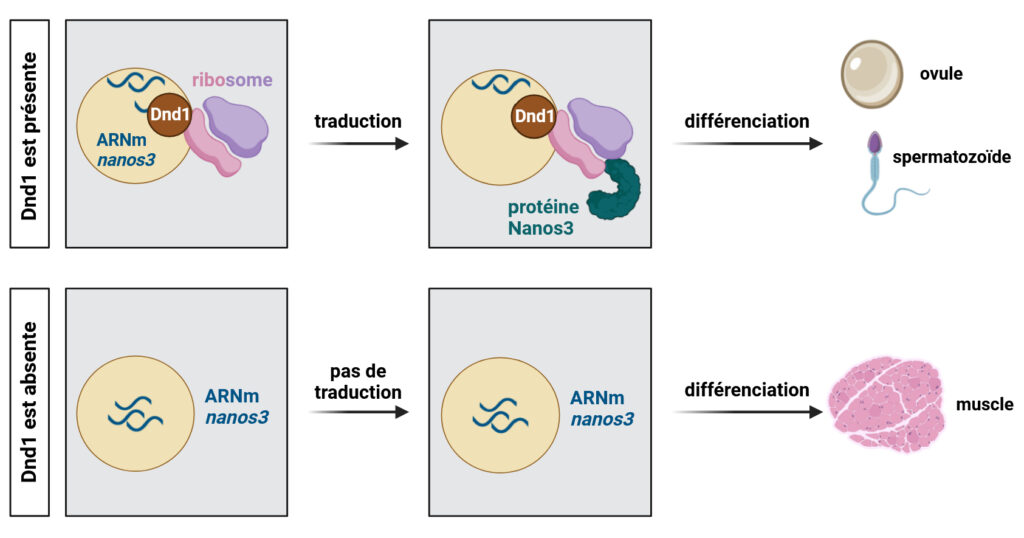
Ainsi, Dnd1 assure un triple rôle : elle relocalise efficacement l’ARNm nanos3, facilite sa traduction en protéine par le ribosome et empêche l’action des microARNs inhibiteurs. Ce processus de traduction est important car la protéine Nanos3 est indispensable pour différencier les cellules non spécialisées en progéniteurs germinaux. Ces futures cellules germinales permettront de transmettre le patrimoine génétique de l’individu à sa descendance, assurant ainsi une continuité au-delà de l’individu.
En absence de Dnd1, les cellules perdent leur capacité à se différencier en cellules germinales, ce qui entraîne une infertilité chez le poisson-zèbre. Les cellules initialement destinées à devenir des spermatozoïdes ou des ovules adoptent alors un destin alternatif et deviennent des cellules musculaires (Figure 3).
Qu’avons-nous appris ?
Cet article met en lumière le rôle crucial de la protéine Dnd1 dans la régulation de l’expression de l’ARNm nanos3 au sein des biocondensats, des structures essentielles au stockage et à la régulation des ARNm nécessaires à la différenciation des précurseurs germinaux chez le poisson-zèbre. Dnd1 permet la localisation de nanos3 à la périphérie des granules, où cet ARNm est traduit en protéine par les ribosomes. La protéine Nanos3 induit ensuite la différenciation des cellules non spécialisées en précurseurs germinaux, garantissant ainsi la transmission de l’information génétique aux générations futures.
La présence de Dnd1 en périphérie du granule est cruciale pour la traduction de la protéine Nanos3, qui est nécessaire pour le maintien du destin germinal de la cellule non différenciée. Toutefois, ce mécanisme de régulation de la traduction des ARNm nanos3 dans les biocondensats n’est pas le seul impliqué. Les chercheurs ont montré que Dnd1 ne contrôle pas tous les gènes impliqués dans la maturation des cellules germinales. La régulation de l’expression de ces gènes est donc faite par un autre intermédiaire.
En révélant le rôle clé de Dnd1 dans la régulation des granules et la différenciation germinale, cette étude met en lumière un mécanisme fondamental qui relie la biologie cellulaire à la continuité de la vie et ouvre la voie à de nouvelles recherches sur les processus de transmission héréditaire. De plus, les biocondensats ne se trouvent pas uniquement dans les cellules germinales, mais aussi dans d’autres types cellulaires. L’étude de ces structures pourrait nous aider à mieux comprendre d’autres mécanismes biologiques fondamentaux.
Éléments pour approfondir
Comment localiser et observer simultanément différents ARNm avec une technique d’hybridation in situ améliorée ? Les ARNm sont des molécules bien plus petites, instables et volatiles que les protéines. Là où visualiser des protéines dans une cellule est relativement simple, la technique ici employée pour regarder simultanément plusieurs ARNm est un peu plus complexe. En effet, il est notamment très compliqué d’observer plusieurs ARNm en même temps, dans une même cellule. Cependant, une technique a été développée en 2011 : le RNAscope. Il s’agit d’un système d’hybridation in situ, c’est-à-dire que l’on marque directement dans la cellule embryonnaire de poisson-zèbre les ARNm grâce à une sonde spécifique [2].
Mais rentrons un peu plus en détail dans cette technique. Le premier objectif est de cibler les ARNm avec une paire de sondes, longues d’environ 20 nucléotides, spécifiques de l’ARNm ciblé (Figure 4).
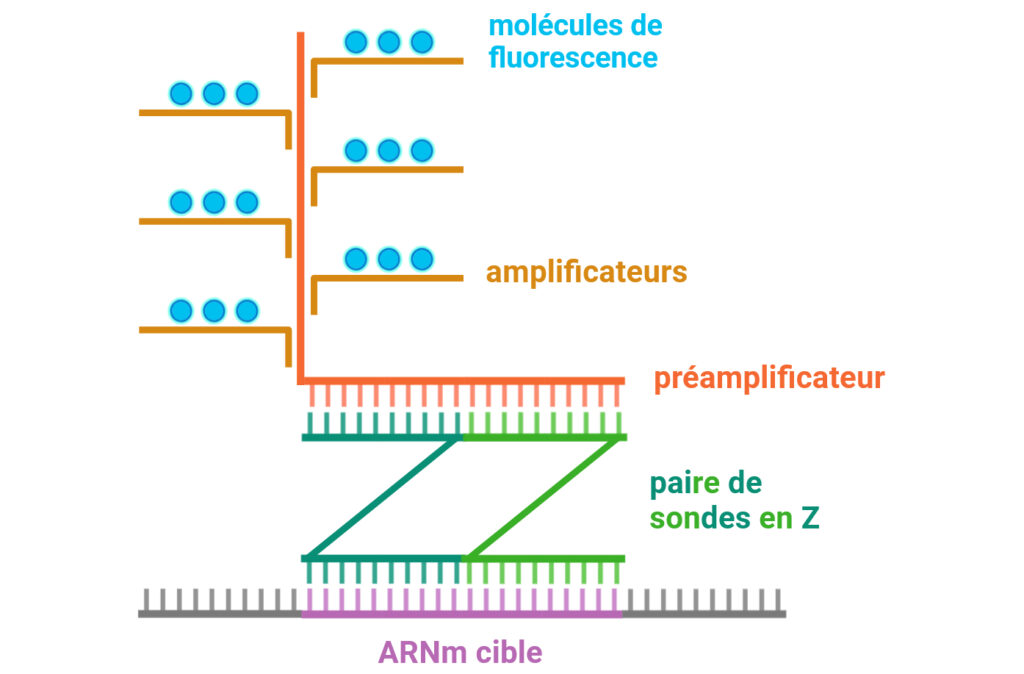
Crédit : adaptée à partir de [3] avec BioRender, usage pédagogique.
Cette paire de sondes, qui possède une forme en Z, se colle aux ARNm cibles et permet ainsi le recrutement de « préamplificateurs ». Ce sont de petites structures qui vont être le support d’autres molécules, les amplificateurs. Ces derniers possèdent une forme particulière, similaire à un arbre. Sur chacune des branches de cet arbre, les amplificateurs portent des molécules de fluorescence (les fluorochromes). Ces molécules peuvent être activées et vont ainsi renvoyer un signal spécifique qui permet la détection, la localisation et la quantification des ARNm ciblés, au sein de la cellule. La grande spécificité de chacune de ces étapes permet de s’assurer que seuls les ARNm cibles sont détectés, ce qui aide à obtenir des résultats fiables.
[1] Cantú A. V., et al., A Pilgrim’s Progress : Seeking Meaning in Primordial Germ Cell Migration. Stem Cell Research, 2017. DOI : 10.1016/j.scr.2017.07.017. [Publication scientifique]
[2] Gross-Thebing T., et al., Simultaneous high-resolution detection of multiple transcripts combined with localization of proteins in whole-mount embryos. BMC Biology, 2014. DOI : 10.1186/s12915-014-0055-7. [Publication scientifique]
[3] Wang F., et al., RNAscope: A Novel in Situ RNA Analysis Platform for Formalin-Fixed, Paraffin-Embedded Tissues. The Journal of Molecular Diagnostics, 2012. DOI : 10.1016/j.jmoldx.2011.08.002. [Publication scientifique]