Chargement de l'article...
Écriture : Kévin Baudry, Benoît Castandet et Bastien Malbert
Relecture scientifique : Juliana Blin et Aurélien Schwob
Relecture de forme : Pauline Colinet et Eléonore Pérès
Temps de lecture : environ 12 minutes.
Thématiques : Génétique & Épigénétique et Microbiologie (Biologie)
Publication originale : Brenner S., et al., An unstable intermediate carrying information from genes to ribosomes for protein synthesis. Nature, 1961. DOI : 10.1038/190576a0

Dans les années 50 et 60, un vent de renouveau commence doucement à souffler sur la société engoncée de l’après-guerre. La brise, d’abord légère, deviendra ouragan et emportera tout sur son passage. Dans la littérature et les arts, la génération Beat pose les bases de ce qui deviendra la contre-culture. Au même moment, la biologie moléculaire vit son âge d’or. Au milieu de cette extraordinaire émulation, des chercheurs unissent leurs efforts pour repousser les limites de la connaissance et posent les fondations, toujours solides, d’une nouvelle discipline. En 1961, une publication fait date par l’importance du résultat et la beauté de la démarche expérimentale. Sydney Brenner, François Jacob et Matthew Meselson vont démontrer l’existence d’une molécule indispensable à l’expression génétique, l’ARN messager.
Le 31 décembre 2019, l’Organisation Mondiale de la Santé recevait de la part des autorités chinoises une étrange carte de vœux. Des cas groupés de pneumonies d’origine inconnue dans la ville de Wuhan intriguent autant qu’ils inquiètent. L’année 2020 pouvait commencer sous les meilleurs auspices. Un an plus tard, tandis que l’épidémie de SARS-CoV-2 a déjà entraîné la mort de plus de deux millions et demi de personnes à travers le monde, des lueurs d’espoir pointent à l’horizon. Alors qu’il faut normalement de nombreuses années pour les développer, plusieurs entreprises pharmaceutiques annoncent l’arrivée sur le marché de leurs vaccins pour début 2021. Un telle rapidité est une première dans l’histoire de la médecine, une révolution ajoutent certains. Ces vaccins sont d’un nouveau genre et très vite les médias font la part belle à une molécule qui jusque-là évoquait plutôt des souvenirs de cours de SVT. Ce sont des vaccins à ARN messager (ARNm).
Qu’est ce qu’un ARN messager ?
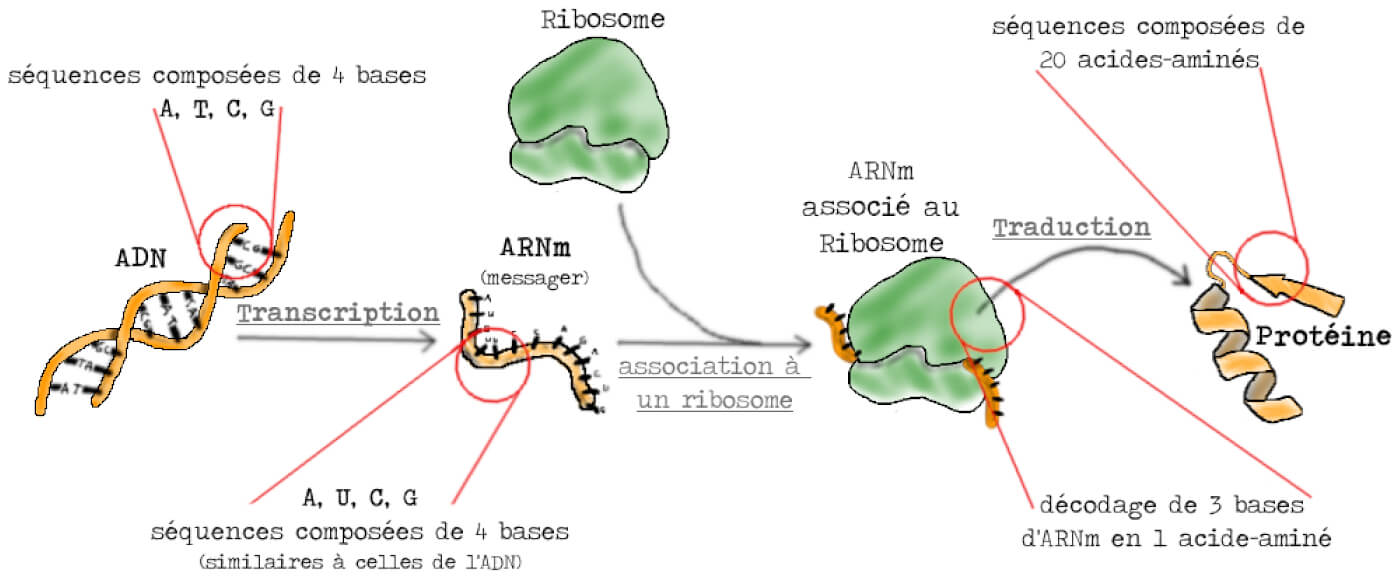
Mais déjà, qu’est ce qu’un ARN ? Un Acide RiboNucléique. Qu’est-ce qu’un messager ? Le porteur d’un message. Un ARN messager serait donc un acide ribonucléique porteur d’un message, nous voilà bien avancés… Pour mieux comprendre, il faut se plonger dans les mécanismes de l’expression génétique. S’il est commun de dire que l’ADN est le support de l’information génétique, une information ne sert pas à grand-chose si elle n’est pas utilisée. L’expression génétique, c’est l’ensemble des mécanismes par lesquels l’information contenue dans les gènes sous forme d’ADN se retrouve exprimée, généralement sous forme de protéines. Celles-ci remplissent de très nombreuses fonctions : elles peuvent aussi bien permettre des réactions biochimiques ou jouer un rôle dans la structure de la cellule. Elles sont synthétisées au niveau des ribosomes, qui peuvent être vus comme des usines à production de protéines. Cette étape est appelée traduction, en référence au fait que les ribosomes traduisent l’information contenue dans l’ADN en protéines.
Et l’ARNm dans tout ça ? Eh bien, c’est un intermédiaire qui transporte l’information génétique des gènes vers les ribosomes, permettant ainsi la synthèse des protéines (Figure 1). À ce propos, quel est le titre du papier que nous mâchons ? « An unstable intermediate carrying information from genes to ribosomes for protein synthesis » soit en français : « Un intermédiaire instable transporte l’information des gènes jusqu’aux ribosomes pour la synthèse des protéines ». S’il est souvent dit qu’un bon titre d’article scientifique doit permettre de comprendre la conclusion des résultats expérimentaux, et ce, en une courte phrase, il faut se rendre à l’évidence : le titre de l’article de Brenner, Jacob et Meselson peut être utilisé comme la définition de l’objet découvert, l’ARNm. Difficile de faire mieux…
Je vous parle d’un temps que les moins… [*]
Pour se rendre compte de l’importance de cette découverte, il faut réaliser que le modèle que nous venons de décrire est encore inconnu dans les années 1950. La décennie qui voit la génération Beat souffler sur les braises de la liberté voit aussi la biologie se transformer profondément. La biologie moléculaire émerge et les découvertes fondamentales s’enchaînent. Bref, elle vit son âge d’or. En 1958, Francis Crick (un des chercheurs à l’origine de la structure de la double hélice de l’ADN) verbalise différentes hypothèses sur les flux d’information entre l’ADN et les protéines. Une de ces hypothèses deviendra célèbre sous le nom de dogme central de la biologie moléculaire et peut être résumée comme suit : une fois que l’information est passée dans la protéine, elle ne peut pas revenir en arrière [1, 2]. Mais comment cette information est-elle transmise de l’ADN à la protéine (Figure 1) ? Il doit bien y avoir un intermédiaire entre l’ADN et les protéines.
Les cerveaux bouillonnent, les idées fusent et les hypothèses fleurissent. Un modèle expérimental en vogue à l’époque est l’infection de la bactérie Escherichia coli par des virus appelés phages (comme par exemple ici). Dans les minutes suivant l’infection par certains phages dits virulents, la synthèse de presque toutes les protéines bactériennes est stoppée. La bactérie devient alors une « sorte de machine » à produire des protéines du phage. En plus des protéines, il est montré qu’un nouveau type d’ARN est également synthétisé. Cet ARN est instable (c’est-à-dire que sa durée de vie est courte) et sa composition ressemble à celle de l’ADN du phage [3]. Et si… et si ce nouvel ARN était l’intermédiaire recherché entre l’ADN et les protéines ? Jacob et Monod lui trouvent un petit nom qui passera à la postérité : ARN messager.
Prendre la route pour trouver des réponses
Toute la question est maintenant de démontrer que cet ARN est un ARNm, c’est-à-dire qu’il permet le transfert de l’information génétique des gènes vers les ribosomes, permettant ainsi la synthèse des protéines. C’est la tâche à laquelle vont s’atteler François Jacob et Sidney Brenner. Ils prennent tous les deux la route pour une épopée vers l’ouest et les grands espaces, à la découverte de l’inconnu. Bon, en fait, ils prennent l’avion et vont rejoindre Matt Meselson à Pasadena, Californie. Nous sommes à l’été 1960.
Ils ont en tête un modèle d’expression génétique qui permettrait d’expliquer les résultats observés lors de l’infection des bactéries par les phages dont nous venons de parler. Dans ce modèle, les ribosomes sont des « usines à produire des protéines » non-spécifiques, capables de synthétiser n’importe quelle protéine à condition d’avoir le bon plan d’assemblage. Le ribosome reçoit l’information génétique des gènes par l’intermédiaire d’un ARNm particulier portant le message du gène. Dans une bactérie infectée par un phage, la synthèse de nouveaux ribosomes et d’ARNm bactériens est interrompue. Les ARNm du virus sont pris en charge par les ribosomes bactériens, qui existaient déjà avant l’infection. Ces ribosomes bactériens synthétisent alors des protéines du virus (quel beau détournement !). Les ARN messagers ont une durée de vie courte (de l’ordre de la minute), ce qui explique que la synthèse de protéines bactériennes s’arrête après l’infection (Figure 2). L’avantage de ce type de modèle, c’est qu’il permet de faire des prédictions qui peuvent ensuite être testées au laboratoire…
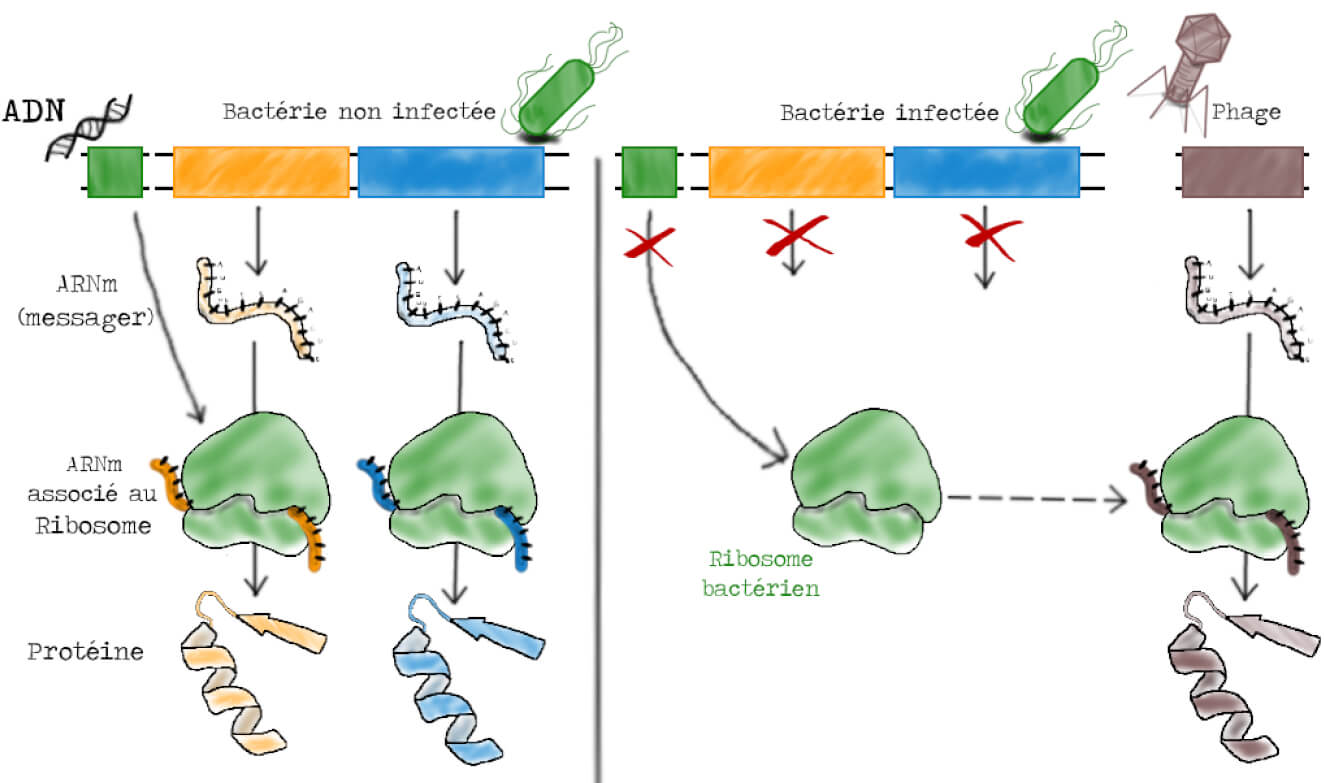
Les prédictions du modèle (Figure 2) sont donc que :
- suite à l’infection par les phages, il n’y a pas de synthèse de nouveaux ribosomes ;
- suite à l’infection par les phages, il y a synthèse d’un ARN instable (à partir de l’ADN du phage) ;
- cet ARN va s’associer aux ribosomes bactériens existant avant l’infection ;
- les protéines du phage vont être synthétisées sur ces mêmes ribosomes.
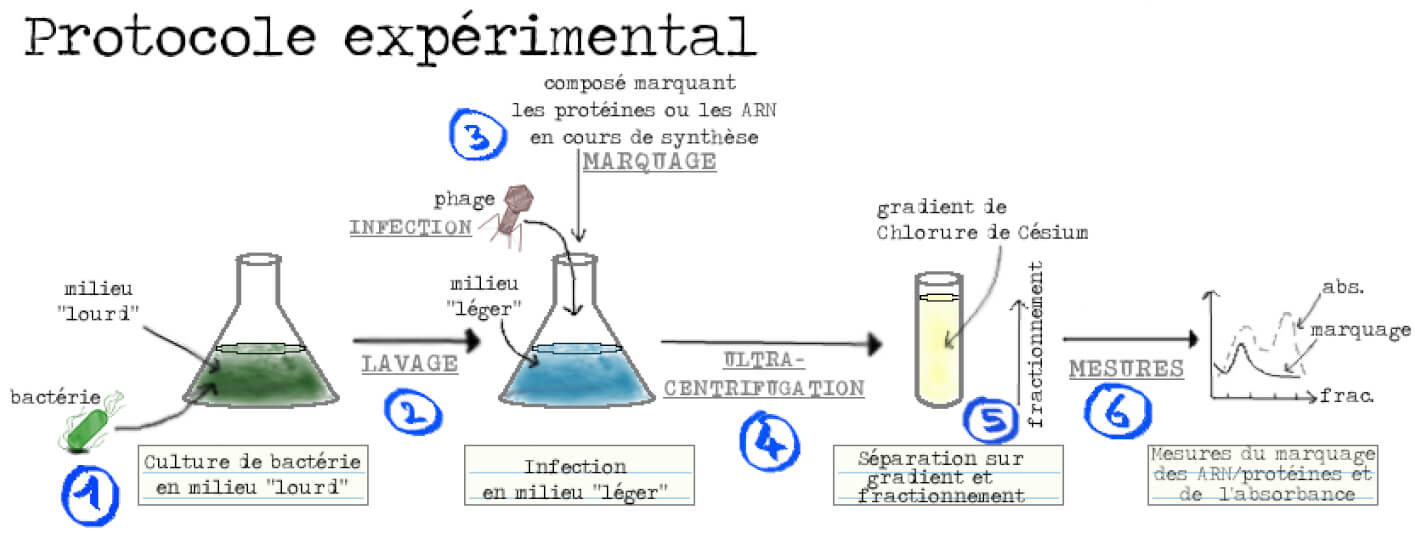
Pour pouvoir tester ces prédictions il faut être capable de déterminer, parmi les composants moléculaires présents, ceux qui avaient été produits avant l’infection de ceux produits après. L’idée géniale des chercheurs a été d’utiliser des isotopes : ce sont des atomes qui ont le même nombre de protons que leur élément de référence mais un nombre de neutrons différent. Par exemple, le carbone 14 (14C) utilisé pour les datations d’objets anciens est un isotope du carbone 12 (12C). On peut donc distinguer des isotopes dits « lourds » qui ont une masse supérieure aux isotopes dits « légers ». Nos chercheurs cultivent alors des bactéries en les nourrissant avec des isotopes lourds, rendant ainsi les différents composants cellulaires, dont les ribosomes, lourds. Les bactéries sont ensuite rincées et placées dans un milieu contenant les isotopes légers et sont en même temps infectées par les phages. Les ribosomes synthétisés après l’infection seront donc composés d’isotopes légers. Il ne reste plus qu’à réaliser une centrifugation à très haute vitesse (ultracentrifugation) afin de séparer les deux types de ribosomes en fonction de leur masse : les ribosomes lourds migrant plus loin (vers le fond du tube) que les légers. Le contenu du tube est ensuite séparé en plusieurs fractions, du fond du tube vers le haut du tube. Une mesure de l’absorbance — c’est-à-dire de l’estimation de la présence et de la concentration d’une substance donnée en solution — sur les fractions issues de la centrifugation permet de localiser celles contenant les ribosomes. Reste à détecter les nouveaux ARN ou protéines dans les différentes fractions, à l’aide d’un marquage, et le tour est joué ! (Figure 3).
Tout ça, c’est de la théorie, il faut maintenant se frotter à la pratique et vérifier que les expériences sont possibles. Il faut d’abord s’assurer qu’il est bien possible de distinguer les ribosomes lourds et légers après centrifugation. Si cela fonctionne, il n’y aura plus qu’à tester le modèle (Figure 4).
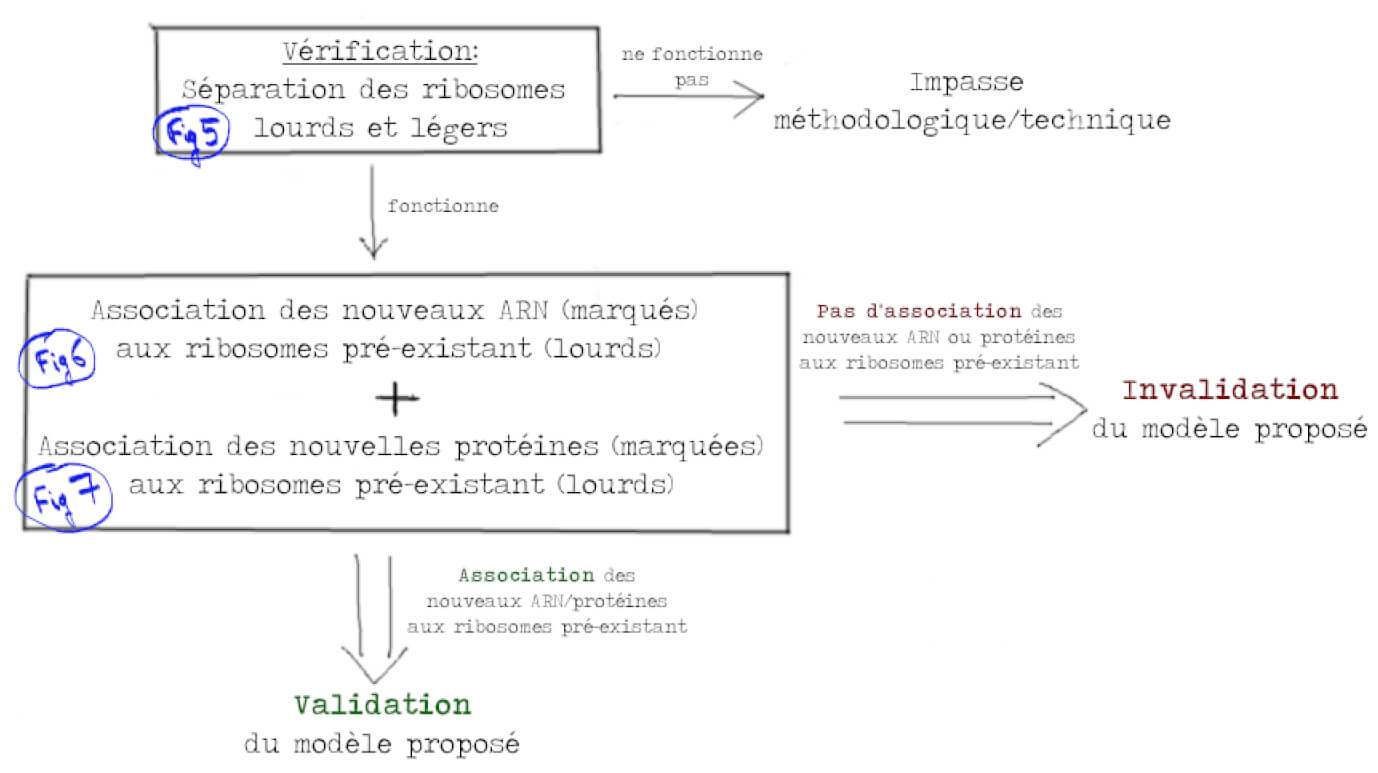
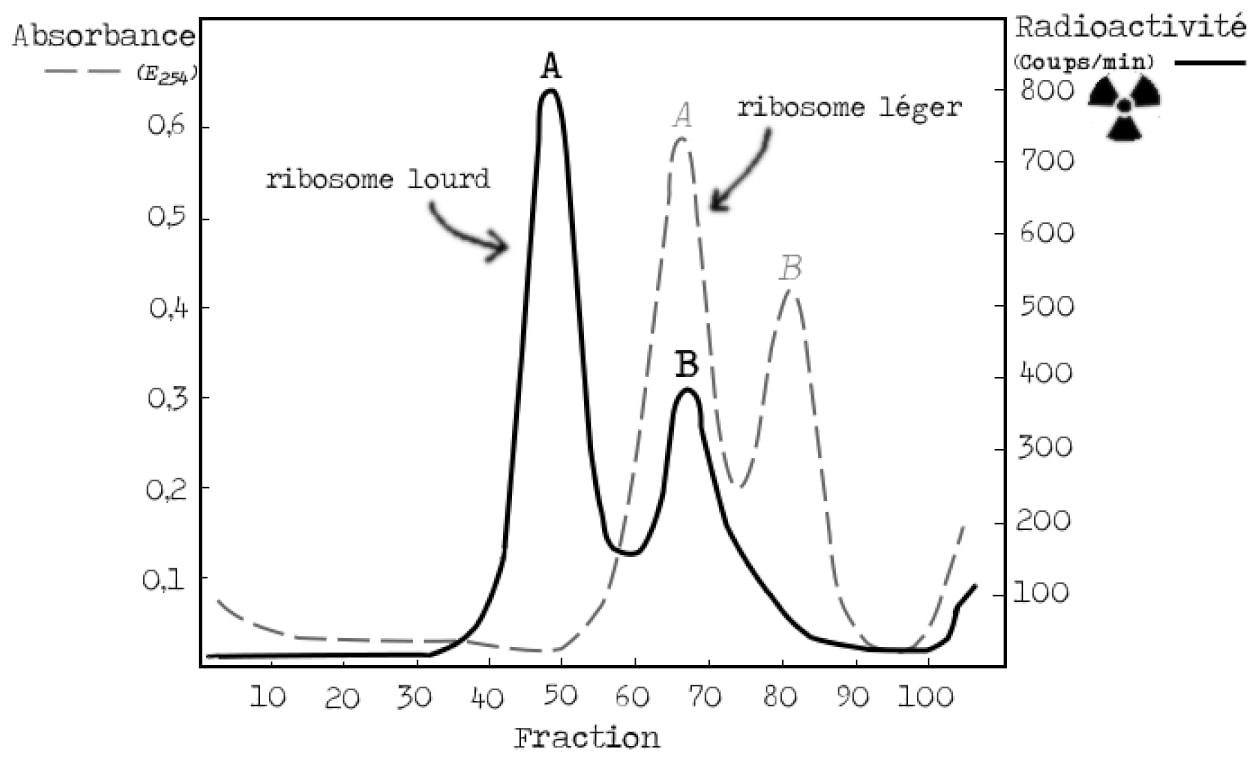
Lors de cette première expérience, il faut donc vérifier que l’on peut séparer les ribosomes lourds des ribosomes légers. On mélange les bactéries « lourdes » avec un excès de bactéries « légères » puis les ribosomes sont séparés. L’excès de ribosomes légers est observé en suivant l’absorbance (ligne en pointillés dans la Figure 5). Comme il n’y a pas assez de ribosomes issus de bactéries lourdes pour pouvoir les observer de la même manière, les chercheurs ont préalablement marqué les ribosomes lourds à l’aide d’un marqueur moléculaire (ligne continue dans la Figure 5). Le résultat est probant, les deux types de ribosomes sont facilement distinguables. Chaque type de ribosome donne deux bandes (dites A et B) correspondant à des états différents et la bande B contient les ribosomes fonctionnels. Détail important, la bande B des ribosomes lourds se superpose avec la bande A des ribosomes légers (Figure 5).
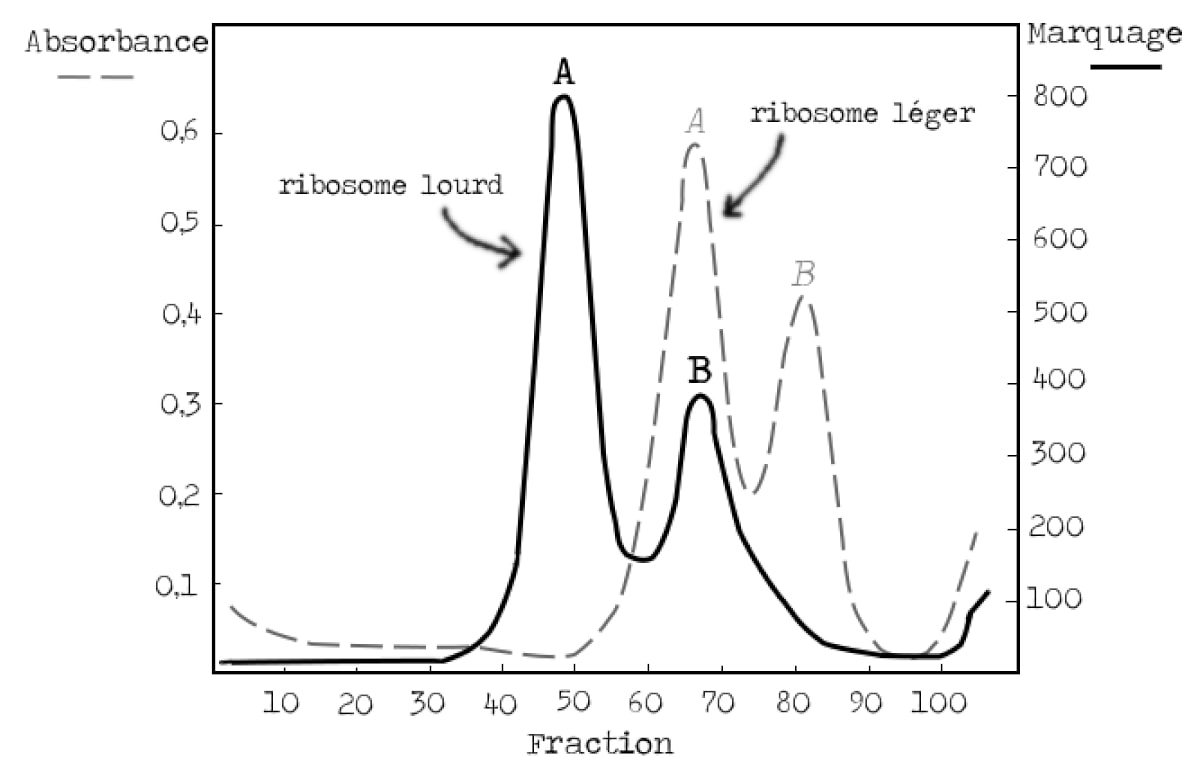
Maintenant que l’on sait séparer les ribosomes lourds et légers, on peut passer à la suite et essayer de valider le modèle (Figure 4). Rappelez-vous, il était connu au moment de l’expérience qu’un nouvel ARN était produit lors de l’infection de la bactérie par le virus. Il faut maintenant montrer que cet ARN s’associe aux ribosomes qui étaient déjà présents avant l’infection. Les bactéries sont donc cultivées en présence d’isotopes lourds, puis infectées par les phages et placées en même temps dans un milieu avec des isotopes légers contenant un marqueur pour suivre la synthèse d’ARN. Elles sont enfin mélangées avec un excès de bactéries infectées cultivées dans un milieu léger (Figure 3). Si l’ARN nouvellement synthétisé s’associe aux ribosomes présents avant l’infection, il se trouvera sur les ribosomes lourds ; autrement son signal sera sur les ribosomes légers (Figure 6).
Les résultats sont sans appel : l’ARN marqué se superpose avec la bande A des ribosomes légers, c’est-à-dire avec la bande B des ribosomes lourds, fonctionnels (Figure 6 et rappel Figure 5). L’ARN produit lors de l’infection est donc ajouté aux ribosomes préexistants. D’autre part, les chercheurs avaient également montré dans une autre expérience que cet ARN est instable (voir version Approfondissement). Suite à l’infection par le phage, nous avons donc production d’un ARN instable qui s’associe aux ribosomes bactériens qui étaient présents avant l’infection.
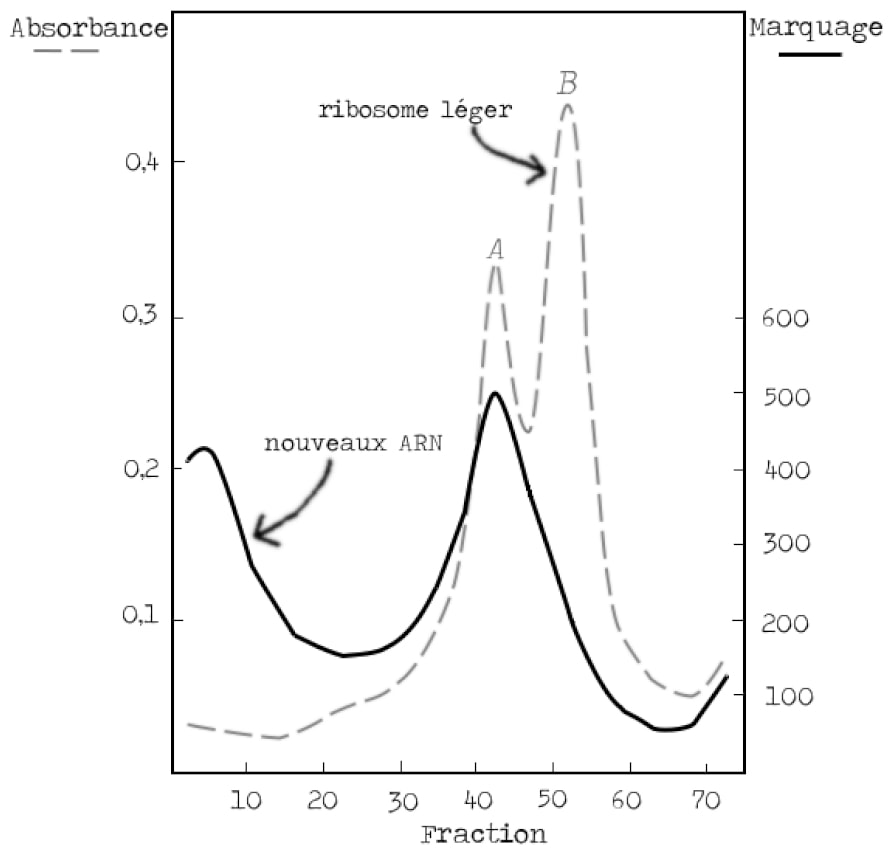
Pour valider le modèle de l’ARN messager, il reste alors à vérifier que les protéines sont synthétisées sur ces mêmes ribosomes bactériens qui étaient présents avant l’infection (Figure 4). Les bactéries cultivées dans un milieu lourd sont incubées avec un marqueur des protéines nouvellement synthétisées au moment de l’infection par le phage. On s’attend à voir un signal correspondant à la synthèse protéique superposé avec la bande B des ribosomes lourds.
Là encore le résultat est sans appel. La bande B des ribosomes lourds se superpose à la bande des protéines marquées, il y a donc synthèse protéique au niveau des ribosomes qui existaient avant l’infection (Figure 7).
L’ARNm, bien plus qu’une hypothèse
Si on fait le bilan des différentes expériences, on se rend compte que suite à l’infection par les phages, un nouvel ARN est synthétisé et s’associe aux ribosomes déjà existants, qui vont produire des protéines. La réunion de tous ces critères est un argument très fort en faveur de l’hypothèse ARNm qui, après un scepticisme initial, sera très vite acceptée de manière généralisée. Comme François Jacob le dit lui-même, non seulement les gens vont accepter l’ARNm, mais beaucoup diront qu’ils l’avaient déjà vu. Il n’empêche que le papier qui est resté dans l’histoire, c’est celui-ci, et pas uniquement pour son résultat. Ce qui est frappant dans cette série d’expériences, c’est l’esthétique de la démarche. Certains vont même jusqu’à dire que c’est l’expérience la plus élégante de l’histoire de la biologie moléculaire. Il faudrait être fou pour vouloir trancher une telle question, il se murmure d’ailleurs que Pâris [**] lui-même trouva plus simple de s’intéresser à la beauté des déesses, on sait où cela l’a mené…
[*] La Bohème, Charles Aznavour
[**] Dans la mythologie grecque, Pâris, fils de Priam roi de Troie, dû désigner la plus belle déesse entre Aphrodite, Athéna et Héra. Il choisit Aphrodite qui lui offre en retour l’amour d’Hélène, la plus belle femme du monde. Or, Hélène était la femme de Ménélas, roi de la cité grecque Sparte. L’enlèvement d’Hélène par Pâris est à l’origine de la mythique Guerre de Troie.
[1] Crick F.H., On protein synthesis. Symposia of the Society for Experimental Biology, 1958. PMID : 13580867. [Publication scientifique]
[2] Jacob F. & Monod J., Genetic regulatory mechanisms in the synthesis of proteins. Journal of Molecular Biology, 1961. DOI : 10.1016/S0022-2836(61)80072-7. [Publication scientifique]
[3] Volkin E. & Astrachan L., Phosphorus incorporation in Escherichia coli ribo-nucleic acid after infection with bacteriophage T2. Virology, 1956. DOI : 10.1016/0042-6822(56)90016-2. [Publication scientifique]
Écriture : Kévin Baudry, Benoît Castandet et Bastien Malbert
Relecture scientifique : Juliana Blin et Aurélien Schwob
Relecture de forme : Pauline Colinet et Eléonore Pérès
Temps de lecture : environ 18 minutes.
Thématiques : Génétique & Épigénétique et Microbiologie (Biologie)
Publication originale : Brenner S., et al., An unstable intermediate carrying information from genes to ribosomes for protein synthesis. Nature, 1961. DOI : 10.1038/190576a0

Dans les années 50 et 60, un vent de renouveau commence doucement à souffler sur la société engoncée de l’après-guerre. La brise, d’abord légère, deviendra ouragan et emportera tout sur son passage. Dans la littérature et les arts, la génération Beat pose les bases de ce qui deviendra la contre-culture. Au même moment, la biologie moléculaire vit son âge d’or. Au milieu de cette extraordinaire émulation, des chercheurs unissent leurs efforts pour repousser les limites de la connaissance et posent les fondations, toujours solides, d’une nouvelle discipline. En 1961, une publication fait date par l’importance du résultat et la beauté de la démarche expérimentale. Sydney Brenner, François Jacob et Matthew Meselson vont démontrer l’existence d’une molécule indispensable à l’expression génétique, l’ARN messager.
Il est des étudiants qui, après avoir longuement usé leur fond de culottes sur les bancs d’une institution d’enseignement supérieur, décident de poursuivre vers une thèse. Ils s’engagent alors pour de nouvelles années de dur labeur et deviennent des chercheurs. Lors de cette formation à la recherche par la recherche, il y a un moment à la fois redouté et attendu. Une étape obligée vers une éventuelle carrière scientifique, un exercice indispensable, presque un rite de passage. Sa simple évocation peut suffire à donner des palpitations aux cœurs les plus placides ou à nouer l’estomac des plus voraces. Non, nous ne parlons pas de la première conférence ou du premier colloque international, ni même de l’écriture du manuscrit de thèse. Nous parlons de l’écriture du premier article scientifique de notre jeune apprenti chercheur.
Comme indiqué ici, la publication scientifique est le moyen pour notre jeune chercheur de partager avec l’ensemble de la communauté les résultats de son travail, de participer au grand effort collectif et millénaire d’édification des connaissances. L’exercice n’est malheureusement pas simple. Il a ses propres codes, ses règles, si bien qu’il existe toute une littérature qui dissèque l’écriture d’un article scientifique, débat de la forme et donne des conseils afin de pouvoir passer cette épreuve. Il y a cependant un point qui semble à peu près faire consensus : le titre est crucial…

Une assertion communément relayée est que l’article est une forme étendue du résumé qui est lui-même une forme étendue du titre. En clair, dans un bon titre, il doit être possible de comprendre la conclusion des résultats expérimentaux, et ce, en une courte phrase. C’est en ce sens que le titre de la publication de Brenner, Jacob et Meselson parue dans Nature en 1961 prend toute sa valeur. Comment définir un ARN messager (ou ARNm) ? Eh bien, on peut dire que c’est un intermédiaire qui porte l’information génétique des gènes vers les ribosomes, ce qui permet d’assurer la synthèse protéique. Quel est le titre de l’article ? « An unstable intermediate carrying information from genes to ribosomes for protein synthesis ». Le titre peut être utilisé comme la définition de l’objet découvert, difficile de faire mieux…
Je vous parle d’un temps que les moins… [*]
Pour se rendre compte de l’importance de cette découverte, il est nécessaire de réaliser que ce que l’on nous apprend aujourd’hui dès le lycée sur le rôle de l’ARN est encore inconnu dans les années 50. Il faut dire que le rôle de l’ADN comme support de l’information génétique fait lui-même encore débat et que les détails ou la question de la réversibilité ou non de la synthèse protéique sont entourés d’un épais brouillard. Bref, au moment où la génération Beat souffle sur les braises de la liberté, la biologie moléculaire vit son âge d’or. Petit flashback : depuis 1944 et des travaux de microbiologie sur les pneumocoques, l’ADN est proposé comme support de l’information génétique [1], résultats confirmés en 1952 en utilisant un modèle sur lequel nous reviendrons, à savoir l’infection des bactéries par les phages [2]. Grâce à des expériences sur le champignon Neurospora crassa, on sait également qu’il existe un lien direct entre les gènes et les protéines, que l’on sait maintenant constitutives des caractères phénotypiques [3]. La compartimentation nucléaire de l’ADN chez les eucaryotes et la possibilité de purifier des fractions cytoplasmiques bactériennes dépourvues d’ADN qui permettent la synthèse de protéines font penser qu’il existerait un intermédiaire nucléotidique assurant le transfert d’information de l’ADN vers le cytoplasme pour la synthèse des protéines. En 1958, Francis Crick présente différentes hypothèses sur les flux d’information entre les acides nucléiques (ADN et ARN) et les protéines dont une version deviendra célèbre sous le nom de dogme central de la biologie moléculaire [4]. L’ARN y joue un rôle pivot entre l’ADN et les protéines. Le dogme central peut être résumé comme suit : une fois que l’information est passée dans la protéine, elle ne peut pas revenir en arrière. Ou reformulé par Jacob et Monod : l’information contenue dans le gène sous la forme ADN est nécessaire et suffisante pour définir la structure de la protéine [5]. La conséquence de ce paradigme est que l’on peut déduire directement la séquence de la protéine à partir de celle du gène.
Les ribosomes étant essentiellement constitués d’ARN, l’hypothèse la plus en vogue à l’époque est que l’intermédiaire dans ce transfert d’information correspond à l’ARN ribosomique (ARNr). On peut résumer ce modèle en disant qu’un gène coderait pour un ARN ribosomique qui permettrait la production d’une protéine (Figure 2).
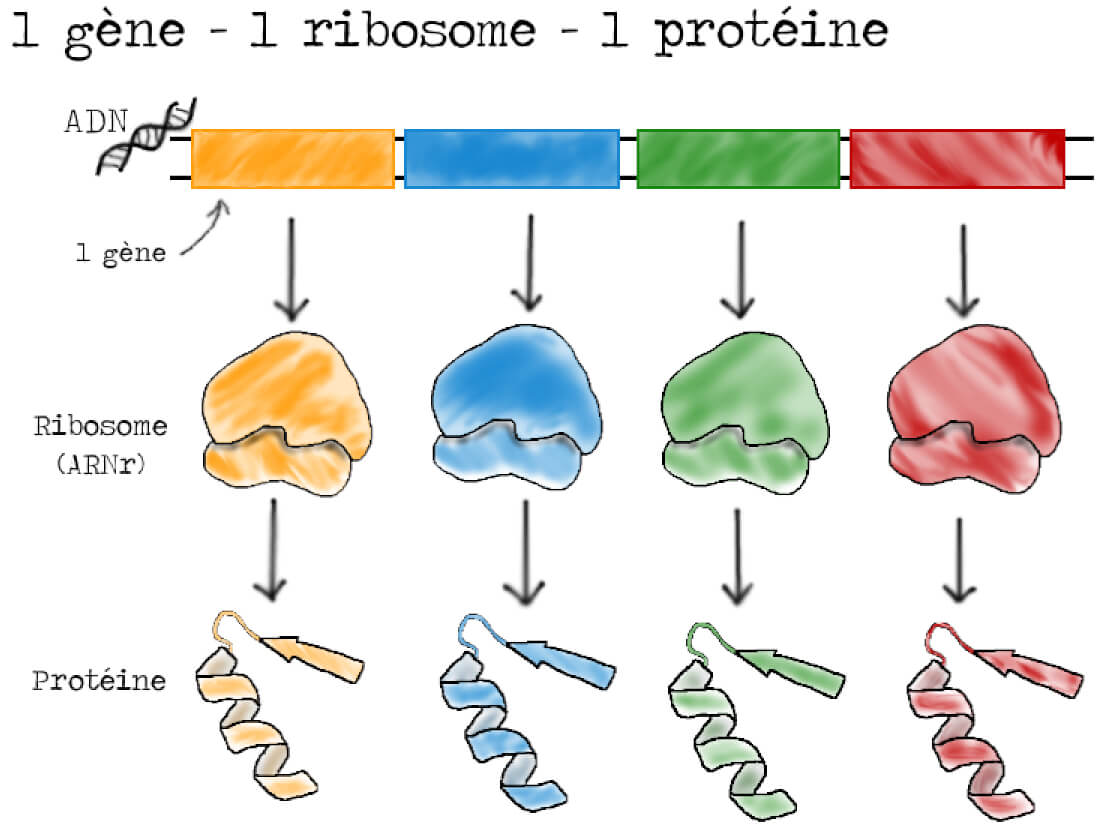
Il y a pourtant des failles dans ce modèle et de plus en plus de résultats expérimentaux le font vaciller. Il est par exemple connu que la composition nucléotidique des ribosomes est très homogène. Comment dès lors expliquer que des gènes différents produisent des ribosomes similaires qui vont ensuite produire des protéines différentes ? Un intermédiaire ARN devrait logiquement refléter la diversité des gènes et donc avoir une composition nucléotidique proche de celle de l’ADN. D’autre part, on sait également que lorsqu’un gène disparaît, la protéine correspondant à ce gène disparaît elle aussi. Cela suggère que l’ARN intermédiaire a un renouvellement rapide et une demi-vie courte alors que les ARN des ribosomes sont plus stables dans le temps (plusieurs jours !).
Des résultats obtenus chez la bactérie Escherichia coli vont accentuer les doutes. Dans les minutes suivant l’infection par un phage (un virus, comme ici) virulent [**], la synthèse de presque toutes les protéines bactériennes est inhibée. La bactérie devient une « sorte de machine » à produire des protéines du virus. En plus des protéines, il est montré qu’un nouveau type d’ARN est également synthétisé. Cet ARN est instable (c’est-à-dire que sa demi-vie est courte) et, propriété importante, sa composition nucléotidique ressemble à celle de l’ADN du phage [6].
Les cerveaux bouillonnent, les idées fusent et les hypothèses fleurissent. Et si… et si ce nouvel ARN était l’intermédiaire recherché entre l’ADN et les protéines ? Oui, mais, comment le montrer ? A priori, trois modèles peuvent expliquer la transformation de la bactérie en machine à produire de la protéine de virus durant une infection. Un de ces modèles fait la part belle à l’existence d’un ARN que la postérité retiendra sous le nom d’ARN messager.
D’abord, les modèles
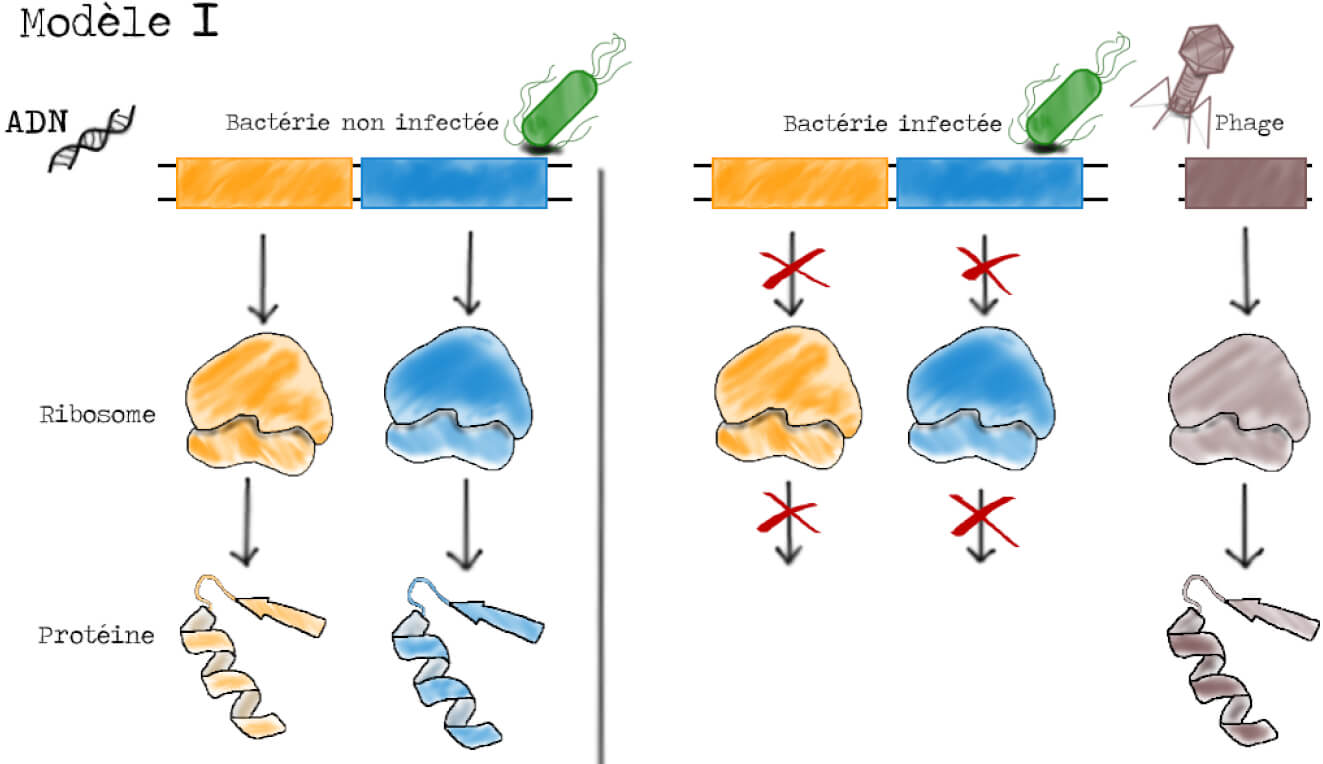
Le premier modèle est le modèle dominant à l’époque, celui du « 1 gène – 1 ribosome – 1 protéine ». Dans une bactérie saine (non-infectée par des phages), l’expression d’un gène donné produit un ribosome spécifique de ce gène qui va ensuite produire la protéine. Lors de l’infection par un phage virulent, la synthèse des ribosomes bactériens est interrompue et de nouveaux ribosomes sont produits à partir des gènes du virus, permettant la synthèse des protéines virales. La présence d’un ARN instable lors de l’infection s’explique si les ARN ribosomiques des phages sont instables (Figure 3).
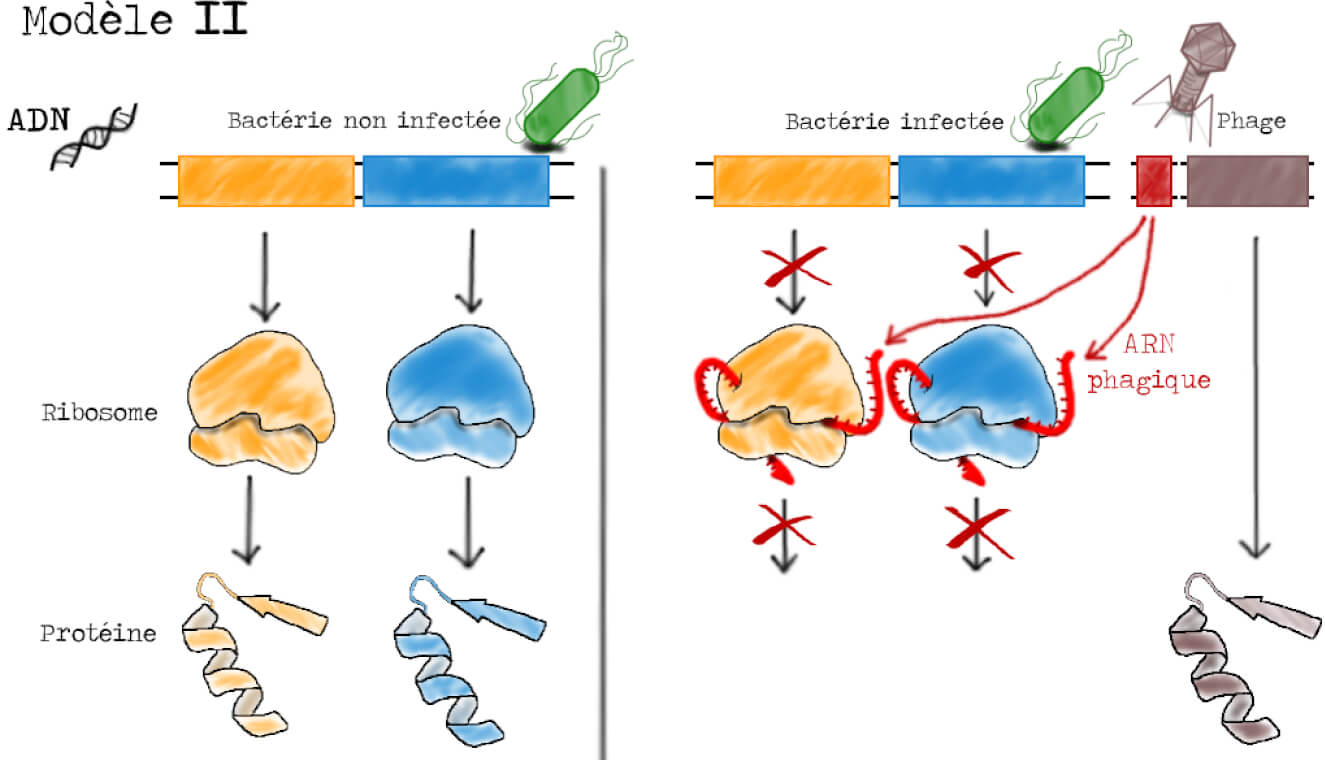
Dans le second modèle, on postule que la synthèse des protéines phagiques et bactériennes ont des mécanismes différents. Dans une bactérie saine, la synthèse protéique a lieu de la même manière que dans le modèle I alors que dans une bactérie infectée, les protéines de phage sont synthétisées directement à partie l’ADN du phage. L’ARN instable produit lors de l’infection a pour rôle d’inhiber la synthèse protéique au niveau des ribosomes bactériens (Figure 4).
Enfin, dans le troisième modèle, qui a les faveurs des auteurs et qu’ils cherchent à démontrer, les ribosomes sont vus comme des « usines à protéines » mais non-spécifiques. Ils sont capables de synthétiser n’importe quelle protéine à condition d’avoir le bon plan d’assemblage. Le ribosome reçoit l’information génétique contenue dans les gènes par l’intermédiaire d’un type d’ARN particulier portant le message du gène. Ce type d’ARN est nommé ARN messager en référence à l’article fraîchement publié de Jacob et Monod où ces derniers théorisent les propriétés de ce type d’ARN, initialement noté « ARN-M » [5]. Dans une cellule infectée, la synthèse de nouveaux ribosomes et d’ARNm bactériens sont interrompus. Les ARNm du phage sont pris en charge par les ribosomes bactériens, existant avant l’infection, qui synthétisent alors des protéines phagiques. Dans ce modèle, ce sont les ARN messagers qui sont instables (Figure 5).
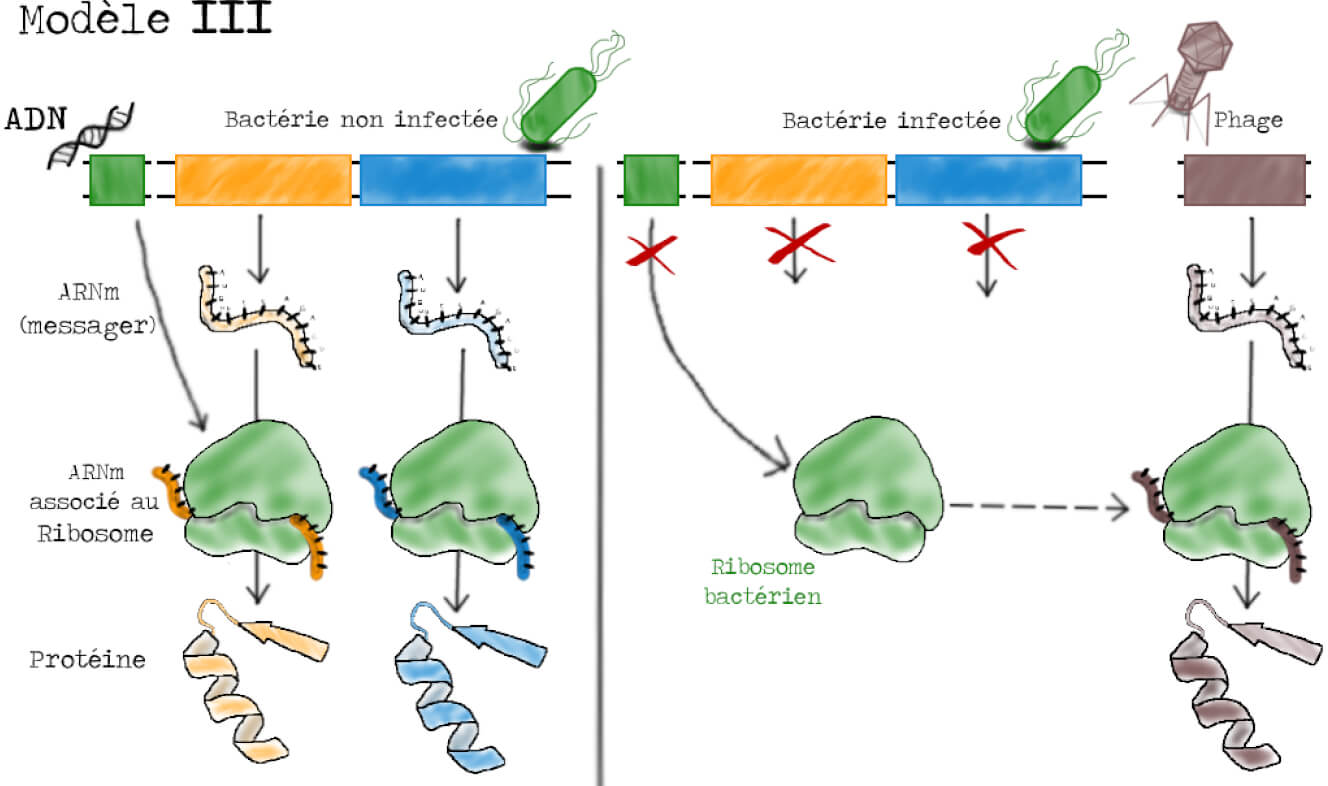
L’immense intérêt de ces trois modèles est qu’ils permettent de faire des prédictions sur le comportement et la localisation des différentes molécules lors de l’infection. Dans le modèle I par exemple, on suppose une production de nouveaux ribosomes suite à l’infection par les phages, ribosomes qui doivent être instables. Dans le modèle II, on suppose une synthèse protéique sans ribosomes, les protéines phagiques ne doivent donc pas se retrouver sur les ribosomes. Enfin, dans le modèle III, on suppose que la synthèse protéique est assurée par des ribosomes préexistant à l’infection.
Cela tombe bien, toutes ces prédictions, il est possible de les tester. Pour prouver l’existence du messager, François Jacob et Sidney Brenner prennent la route (et accessoirement l’avion) direction l’ouest et la Californie. Ils vont travailler avec Matt Meselson (oui oui, le fameux Meselson de la réplication semi conservative de l’ADN ! [7]) dans le laboratoire de Max Delbrück à Pasadena. Nous sommes à l’été 1960.
Puis, le protocole
Afin de trancher entre les différents modèles, il est essentiel de pouvoir discriminer les ribosomes présents avant l’infection de ceux produits après. L’idée géniale est d’utiliser des isotopes : ce sont des atomes qui ont le même nombre de protons que leur élément de référence mais un nombre de neutrons différent. Les molécules organiques contenant les isotopes 15N et 13C (isotopes dits « lourds ») de l’azote et du carbone auront donc une masse plus grande que celles contenant les isotopes de référence 14N et 12C (dits « légers »).
Nos chercheurs font pousser des bactéries en les nourrissant avec des isotopes lourds. Tous les composants cellulaires seront donc lourds et a fortiori les ribosomes. Les bactéries sont ensuite rincées et placées dans un milieu contenant les isotopes légers et sont en même temps infectées par le phage. Les ribosomes synthétisés après l’infection seront donc, eux, légers. Il ne reste plus qu’à réaliser une ultracentrifugation afin de séparer les deux types de ribosomes. Une mesure de l’absorbance sur les fractions issues de la centrifugation permet de localiser celles contenant les ribosomes. Si, au moment de l’infection par les phages, on ajoute en plus des nucléotides ou des acides aminés radioactifs, il devient possible de suivre l’apparition de nouveaux ARN ou protéines car ces molécules seront radioactives. On peut donc distinguer et suivre deux choses : léger ou lourd (pour les ribosomes), radioactif ou non (pour les ARN ou les protéines) (Figure 6).
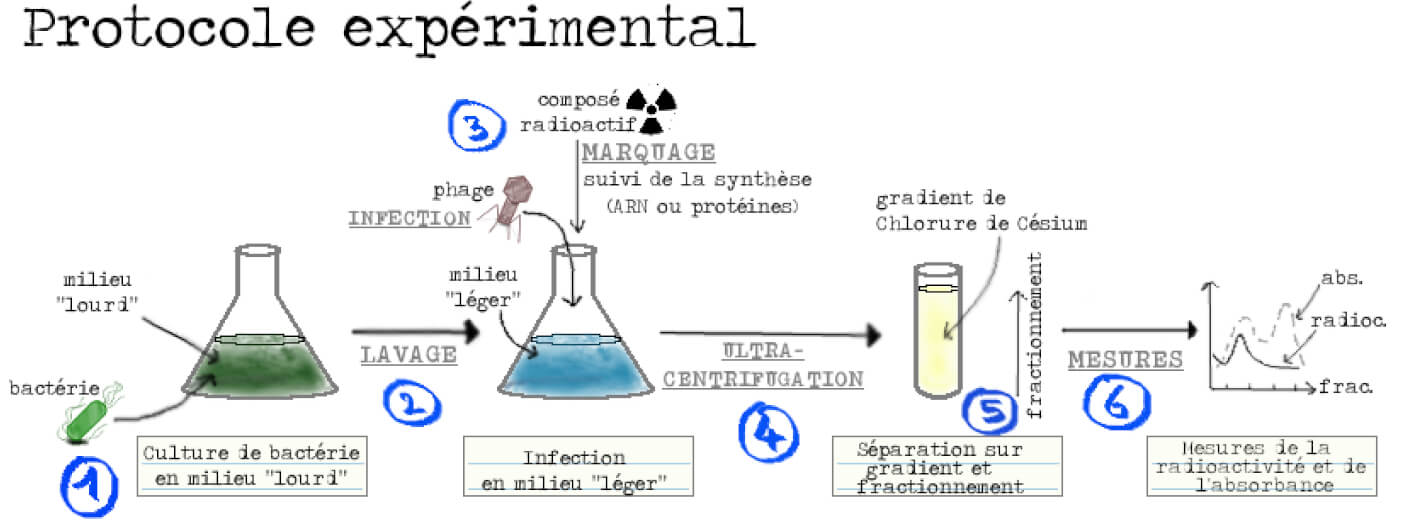
Ensuite, quelques contrôles
Tout ça c’est de la théorie, il faut maintenant vérifier que les expériences sont réalisables. Le premier contrôle est de s’assurer qu’il est bien possible de distinguer les ribosomes lourds et légers après centrifugation. Pour cette expérience contrôle, les ribosomes lourds sont en plus marqués radioactivement grâce à l’ajout dans le milieu de culture de phosphate radioactif. On mélange les bactéries « lourdes » avec un excès de bactéries « légères » et les ribosomes sont séparés. L’excès de ribosomes légers est observé en suivant l’absorbance et les ribosomes lourds par suivi de la radioactivité. Le résultat est probant, les deux types de ribosome sont facilement distinguables. Chaque ribosome donne deux bandes (dites A et B) correspondant à des niveaux d’assemblage différents des ribosomes (les ribosomes fonctionnels sont dans la bande B) et, détail important, la bande B des ribosomes lourds colocalise (= se superpose) avec la bande A des ribosomes légers (Figure 7).
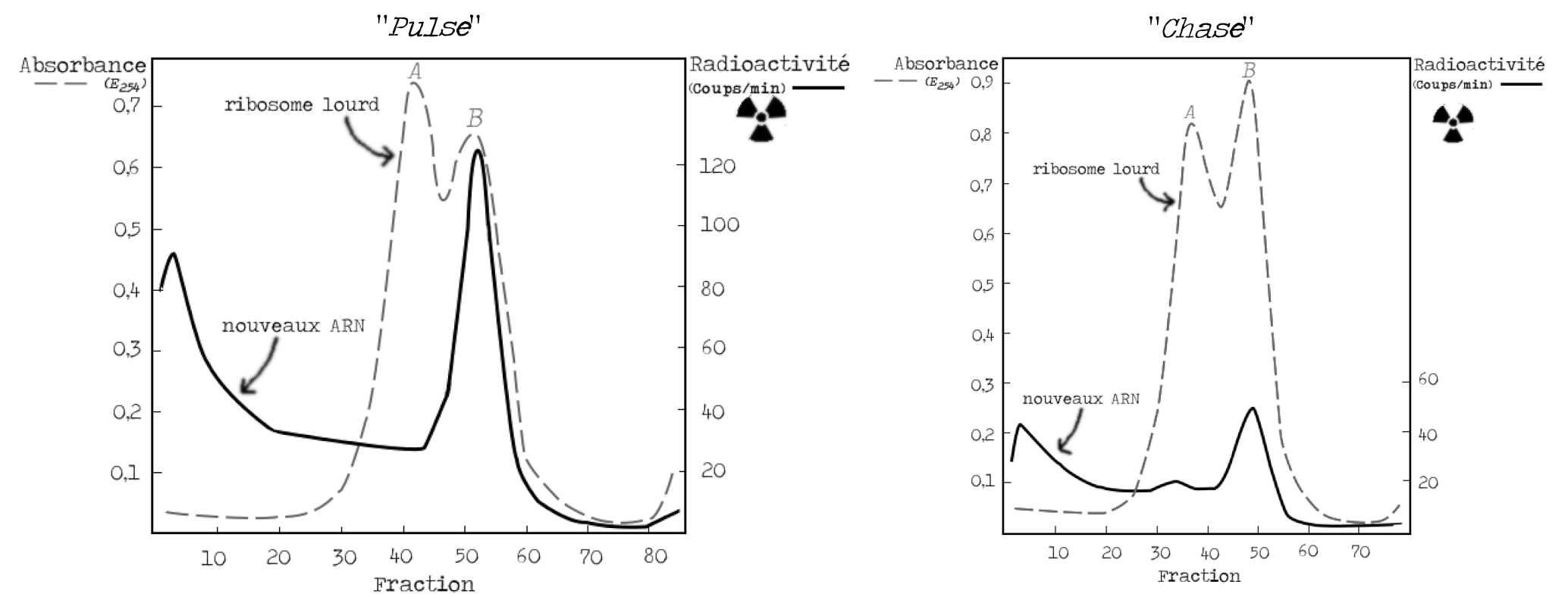
Le second contrôle consiste à vérifier qu’il est bien possible de mettre en évidence la présence d’un ARN instable lors de l’infection par le phage. Le moyen le plus approprié de le montrer est une expérience de pulse-chase. Au moment de l’infection par le phage, un précurseur radioactif d’ARN est rajouté dans le milieu de culture pendant un bref laps de temps (« pulse »). Tous les ARN nouvellement synthétisés seront donc radioactifs. On ajoute ensuite un excès de précurseur non radioactif (« chase ») et on suit le devenir du marquage radioactif. Les molécules instables, à la demi-vie courte, vont voir leur marquage disparaître. C’est exactement ce qu’il se passe. Un ARN radioactif est visible au niveau de la bande B des ribosomes et le marquage radioactif diminue lors de la chase (Figure 8).
Pour résumer : il est possible de séparer les ribosomes lourds des ribosomes légers et il est également possible de suivre un ARN instable qui se trouve au niveau de la bande B des ribosomes. Tout est réuni pour pouvoir tester les trois modèles.
Enfin, l’épreuve du réel
La première étape est de tester le modèle I (Figure 3). Comme nous l’avons vu, celui-ci implique que de nouveaux ribosomes sont synthétisés au moment de l’infection et qu’une partie au moins des ARN du ribosome est instable. Les bactéries sont donc cultivées en présence d’isotopes lourds, puis infectées et placées dans un milieu avec des isotopes légers contenant du phosphate radioactif pour suivre la synthèse d’ARN, et enfin mélangées avec un excès de bactéries infectées cultivées dans un milieu léger (Figure 9). Si de nouveaux ribosomes sont synthétisés au moment de l’infection, ils seront radioactifs ET légers.
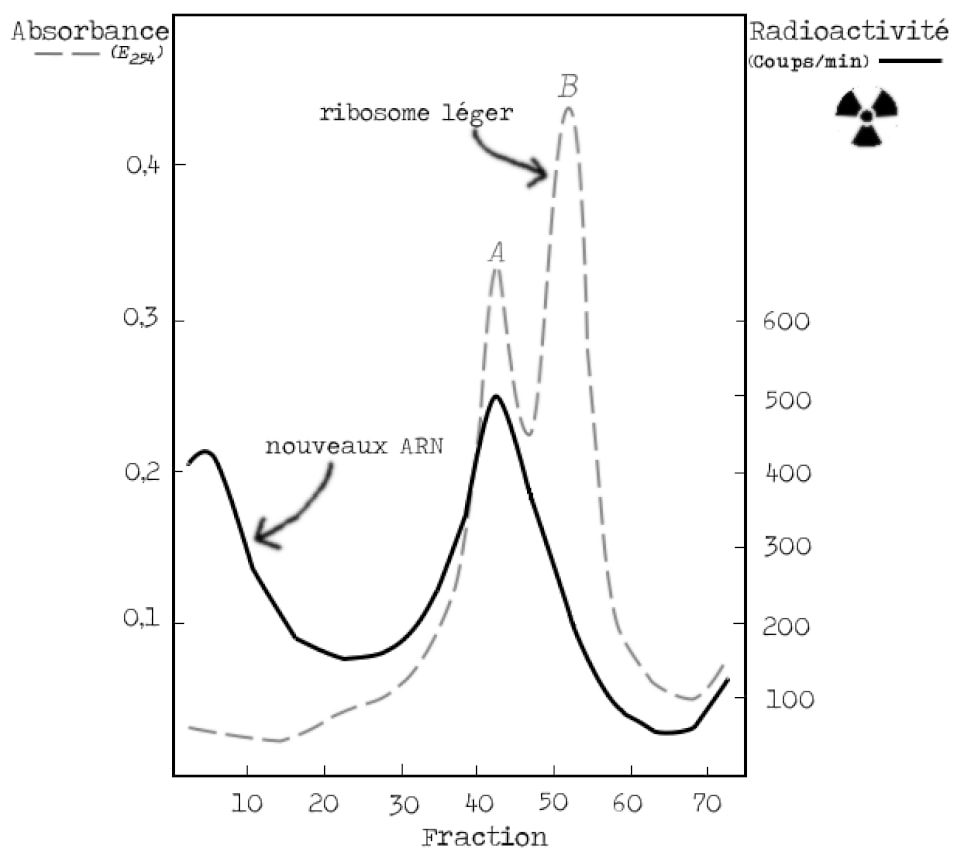
Les résultats sont sans appel : l’ARN marqué localise avec la bande A des ribosomes légers, c’est-à-dire avec la bande B des ribosomes lourds (pour rappel, voir Figure 7). Il n’y a donc pas de formation de nouveaux ribosomes, le modèle I est abandonné. L’ARN produit lors de l’infection est ajouté aux ribosomes préexistants.
Pour l’instant, les modèles II et III tiennent encore debout et sont toujours en course. L’ARN identifié sur les ribosomes lourds peut en effet être le messager recherché (modèle III, Figure 5) ou l’ARN phagique qui va inactiver ces ribosomes (modèle II, Figure 4). La prédiction fondamentale du modèle II est que la synthèse des protéines du phage se fait directement sur l’ADN phagique, on ne devrait donc pas observer de synthèse protéique sur les ribosomes. Afin de trancher, les bactéries cultivées dans un milieu lourd sont incubées avec un précurseur radioactif de la synthèse protéique (35S) (soufre 35) au moment de l’infection par le phage (Figure 10). Si le modèle III est correct, on s’attend à voir de la synthèse protéique (un signal radioactif) qui se superpose à la bande B des ribosomes lourds. Un tel signal doit être absent dans le cadre du modèle II.
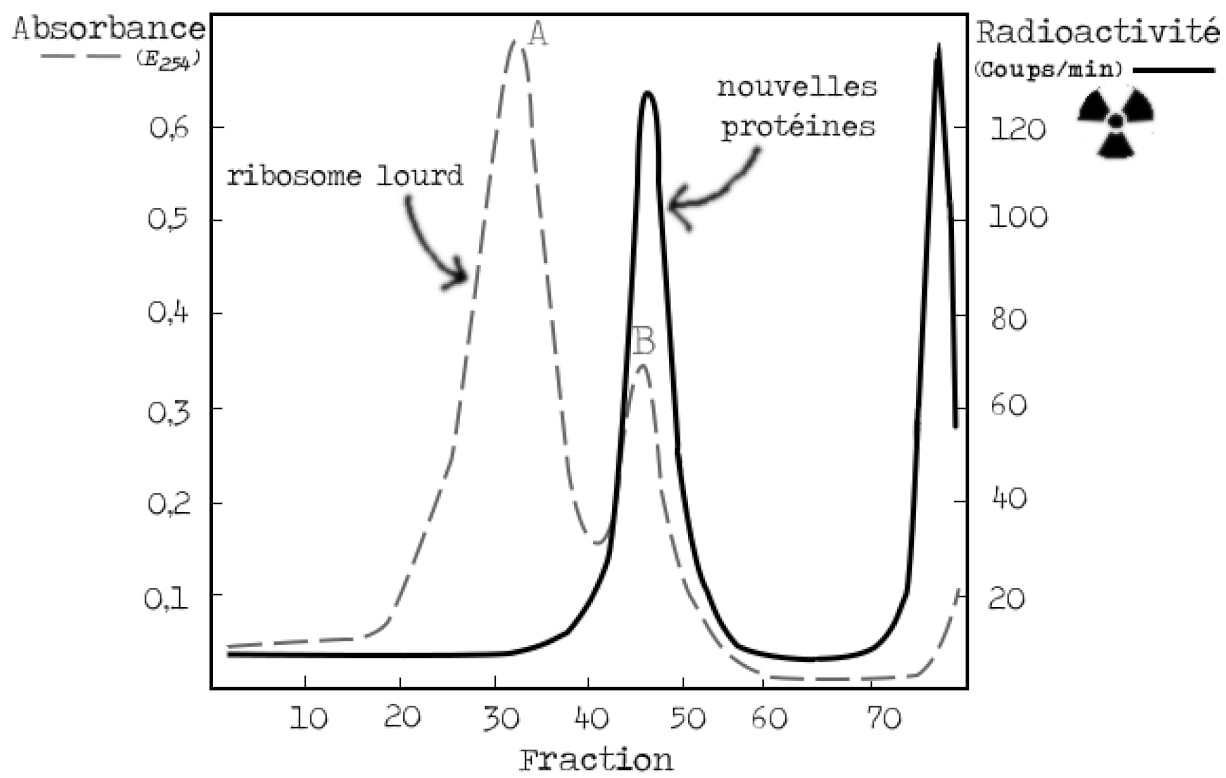
Là encore le résultat est sans appel. La bande B des ribosomes lourds est marquée radioactivement, il y a donc synthèse protéique au niveau des ribosomes qui existaient avant l’infection.
Et le gagnant est…le modèle III
Si on fait le bilan des différentes expériences, on se rend compte qu’il n’y a pas synthèse de nouveaux ribosomes suite à l’infection par les phages, qu’un nouvel ARN est par contre synthétisé et qu’il s’associe aux ribosomes déjà existants qui vont produire des protéines. Le seul modèle compatible avec l’ensemble de ces résultats est le modèle III. Il existe donc un ARN messager.
Très vite, le scepticisme de la communauté va faire place à une acceptation généralisée. Comme Jacob le dit lui-même, non seulement les gens vont accepter le messager, mais beaucoup diront qu’ils l’avaient déjà vu. Il n’empêche que le papier qui est resté dans l’histoire, c’est celui-ci, et pas uniquement pour son résultat. Ce qui est frappant dans cette série d’expériences, c’est l’esthétique de la démarche. Certains vont même jusqu’à dire que c’est l’expérience la plus élégante de l’histoire de la biologie moléculaire. Il faudrait être fou pour vouloir trancher une telle question, il se murmure d’ailleurs que Pâris [***] lui-même trouva plus simple de s’intéresser à la beauté des déesses, on sait où cela l’a mené .
La beauté vient certainement du rôle des modèles dans la mise au point des expériences. Ils permettent de faire des prédictions sur l’existence et le comportement de certaines molécules et ce sont ces prédictions qui sont testées expérimentalement. C’est ce que certains appellent la « théorie Figure 1 » [8] : on commence par poser la question biologique sous forme de modèle, il est la première Figure de l’article. En somme, une parfaite illustration de ce que l’on nous apprend dès l’école à propos de la méthode scientifique. Enfin, à l’heure où certains prophétisent que la révolution des approches expérimentales à haut débit pourrait bientôt permettre à la biologie de se passer d’hypothèses [9], le parfum de nostalgie qui peut se dégager d’un tel papier n’est sûrement pas étranger à sa beauté.
Pour aller plus loin :
On conseille le visionnage d’une interview de François Jacob réalisée par Michel Morange. Les vignettes 40 à 43 traitent de la découverte de l’ARN messager. Le rôle du magnésium n’aura plus de secret pour vous.
De la même manière, François Jacob consacre de longs passages à cette découverte dans son livre « La statue intérieure » disponible en poche chez Folio.
[*] La Bohème, Charles Aznavour.
[**] Un virus virulent conduit à un cycle lytique. Une infection virale peut conduire soit à un cycle lytique au cours duquel la réplication précède la sortie de virions de la cellule, soit à un cycle lysogénique au cours duquel le génome viral s’intègre au génome cellulaire. À noter que les deux cycles peuvent se succéder lors d’une même infection.
[***] Dans la mythologie grecque, Pâris, fils de Priam roi de Troie, dû désigner la plus belle déesse entre Aphrodite, Athéna et Héra. Il choisit Aphrodite qui lui offre en retour l’amour d’Hélène, la plus belle femme du monde. Or, Hélène était la femme de Ménélas, roi de la cité grecque Sparte. L’enlèvement d’Hélène par Pâris est à l’origine de la mythique Guerre de Troie.
[1] Avery O.T., et al., Studies on the Chemical Nature of the Substance Inducing Transformation of Pneumococcal Types : Induction of Transformation by a Desoxyribonucleic Acid Fraction Isolated from Pneumococcus Type III. The Journal of Experimental Medicine, 1944. DOI : 10.1084/jem.79.2.137. [Publication scientifique]
[2] Hershey A.D. & Chase M., Independent functions of viral protein and nucleic acid in growth of bacteriophage. The Journal of general physiology, 1952. DOI : 10.1085/jgp.36.1.39. [Publication scientifique]
[3] Beadle G.W. & Tatum E.L., Genetic Control of Biochemical Reactions in Neurospora. PNAS, 1941. DOI : 10.1073/pnas.27.11.499. [Publication scientifique]
[4] Crick F.H., On protein synthesis. Symposia of the Society for Experimental Biology, 1958. PMID : 13580867. [Publication scientifique]
[5] Jacob F. & Monod J., Genetic regulatory mechanisms in the synthesis of proteins. Journal of Molecular Biology, 1961. DOI : 10.1016/S0022-2836(61)80072-7. [Publication scientifique]
[6] Volkin E. & Astrachan L., Phosphorus incorporation in Escherichia coli ribo-nucleic acid after infection with bacteriophage T2. Virology, 1956. DOI : 10.1016/0042-6822(56)90016-2. [Publication scientifique]
[7] Meselson M. & Stahl F.W., The Replication of DNA in Escherichia Coli. PNAS, 1958. DOI : 10.1073/pnas.44.7.671. [Publication scientifique]
[8] Phillips R., Theory in Biology: Figure 1 or Figure 7? Trends in cell biology, 2015. DOI : 10.1016/j.tcb.2015.10.007. [Publication scientifique]
[9] Mazzocchi F., Could Big Data be the end of theory in science? EMBO Reports, 2015. DOI : 10.15252/embr.201541001. [Publication scientifique]